24082010 Python processing
pythonでprocessingっぽいことをやろうというプロジェクトが立ち上がってた。
楽しみ。
bruceとかでかっこいいプレゼンが出来るようになると嬉しい。
24082010 Python processing
23082010 life
電子書籍が普及しまくったら図書館ってどういう位置づけになるの?とかそういう疑問があって電子書籍やビジネスに関する本なども読んでいる。
というより、論文がpdf化している割には使いづらいのが気に入らない。doiとかつけといてアトミックな単位がそこか?とか思っている。例えばReal World Haskellみたいにパラグラフ毎にコメントを入れられるようにして、フリーズした状態じゃなくてもっと流動的な知識のストレージの受け皿みたいなのが、必要なんじゃないかなぁと。そういったCGM的なインフラって出来ないもんかね?
18082010 chemoinformatics Python network igraph
MCSを求めるには比較したい二つの化合物のModular Productを求めて、それの最大クリーク探索をすればいい。
Modular Productとは
にエッジを張ったグラフ。
これを作ってあとはigraphの最大クリーク探索メソッドで
import openbabel as ob
from igraph import *
sm1 = 'OCC(N)=O'
sm2 = 'O=CC(N)=O'
obc = ob.OBConversion()
obc.SetInAndOutFormats('smi','smi')
source = ob.OBMol()
obc.ReadString(source,sm1)
source_atoms = [a for a in ob.OBMolAtomIter(source)]
target = ob.OBMol()
obc.ReadString(target,sm2)
target_atoms = [a for a in ob.OBMolAtomIter(target)]
pairs = []
for i in range(len(source_atoms)):
for j in range(len(target_atoms)):
if source_atoms[i].GetAtomicNum() == target_atoms[j].GetAtomicNum():
pairs.append((source_atoms[i].GetIdx(),target_atoms[j].GetIdx()))
p = []
for x in range(len(pairs)-1):
for y in range(x+1,len(pairs)):
u = pairs[x]
v = pairs[y]
if u[0] != v[0] and u[1] != v[1]:
sb = source.GetBond(u[0],v[0])
tb = target.GetBond(u[1],v[1])
if sb != None: sbo = sb.GetBondOrder()
if tb != None: tbo = tb.GetBondOrder()
if (sb == None and tb == None) or (sb != None and tb != None and sbo == tbo):
p.append((pairs.index(u),pairs.index(v)))
g = Graph(p)
mc = g.largest_cliques()
for c in mc:
print [pairs[i] for i in c]
実際にはline graph(エッジとノードを入れ替えたグラフ)のModular Productをもとめてクリーク探索したほうが計算量がかなり減るらしい。
が、今回はやっていない。
 Handbook of Chemoinformatics Algorithms (Chapman & Hall/Crc Mathematical and Computational Biology)
Handbook of Chemoinformatics Algorithms (Chapman & Hall/Crc Mathematical and Computational Biology) Chemoinformatics and Computational Chemical Biology (Methods in Molecular Biology)
Chemoinformatics and Computational Chemical Biology (Methods in Molecular Biology)igraphはRとPythonのbindingがあるのにチュートリアルがほとんどなさげなので、ネットワーク分析の本があると手っ取り早く知れて良い。
クリーク探索
こんなネットワークからクリークを探してみる
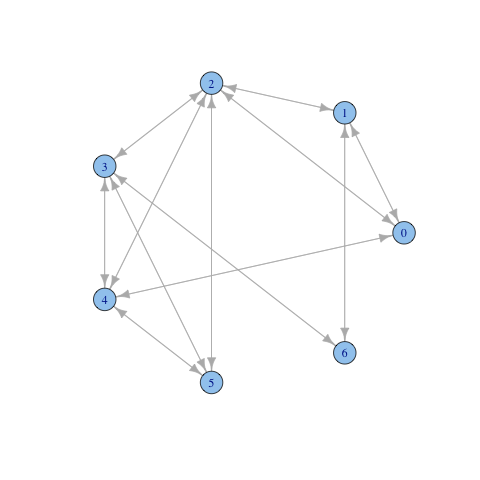
g <-matrix(c(0,1,1,0,1,0,0,1,0,1,0,0,0,1,1,1,0,1,1,1,0,0,
0,1,0,1,1,1,1,0,1,1,0,1,0,0,0,1,1,1,0,0,0,1,
0,1,0,0,0),nrow=7,ncol=7,byrow=TRUE)
a <- graph.adjacency(g)
maximal.cliques(a)
[[1]]
[1] 0 1 2
[[2]]
[1] 1 6
[[3]]
[1] 0 2 4
[[4]]
[1] 2 3 4 5
[[5]]
[1] 3 6
Pythonの場合
a = [[0,1,1,0,1,0,0],
[1,0,1,0,0,0,1],
[1,1,0,1,1,1,0],
[0,0,1,0,1,1,1],
[1,0,1,1,0,1,0],
[0,0,1,1,1,0,0],
[0,1,0,1,0,0,0]]
g = GraphBase.Adjacency(a,mode=ADJ_UNDIRECTED)
g.maximal_cliques() # [(0, 1, 2), (1, 6), (0, 2, 4), (2, 3, 4, 5), (3, 6)]
g.largest_cliques() # [(2, 3, 4, 5)]
17082010 drum'n'bass
hospitalのpodcast聴いてて amen alley /danny byrdやばすぎ!とか思ったら、すでにyoutubeにあがっていた。
これいつリリースされんだろかね。
昔のでも聴いとくか。
16082010 chemoinformatics Python cytoscape
MCSSTanimotoで組んだネットワークのメリットはなんといってもエッジの属性にMCSの情報(smilesとかInChi)をのっけられることであろう。
import sys
from Gaston import MCSSTanimoto,Gaston
import openbabel as ob
obc = ob.OBConversion()
obc.SetInAndOutFormats("sdf", "smi")
input = "pc_sample.sdf"
mol = ob.OBMol()
next = obc.ReadFile(mol,input)
mols = [mol]
while next:
mol.StripSalts(14)
mols.append(mol)
mol = ob.OBMol()
next = obc.Read(mol)
mols = mols[1:11]
for i in range(len(mols)-1):
for j in range(i+1,len(mols)):
sys.stdout.flush()
mcs = MCSSTanimoto(mols[i],mols[j])
if mcs.score() > 0.2:
print "%s\t%s\t%s\t%s\t%2.3f" % (mols[i].GetTitle(), "mcs", \
mols[j].GetTitle(), obc.WriteString(mcs.mcs).split()[0], mcs.score())
でも、実際に組んでみたら、解釈が意外に難しそうなことに気がついた。MCSSTanimotoのアルゴリズム自体が問題なのかもしれない。
MCSよりはmolblaster的に細切れにしてみて最頻出のフラグメントからネットワークを組んでいくか、ありがち置換基の定義済みテーブルを使って類似性の高い置換基動どうしをつなぐという経験ベースのアプローチのほうが直感的かもしれないなぁ。
15082010 chemoinformatics java
GastonでMCSを探索するのは色々無理があったようで、どうするかな困った困ったと探していたら、SMSDを見つけた。cdkには既に組み込まれているらしくてさっきビルドしてみたんだけど、よく考えたらCDKの使い方あんまよくわかんないや、、、ってことでGUIで遊んだ。
ちなみに、java6でビルドしないといけないので、osx(10.5)の場合は環境変数を設定した。
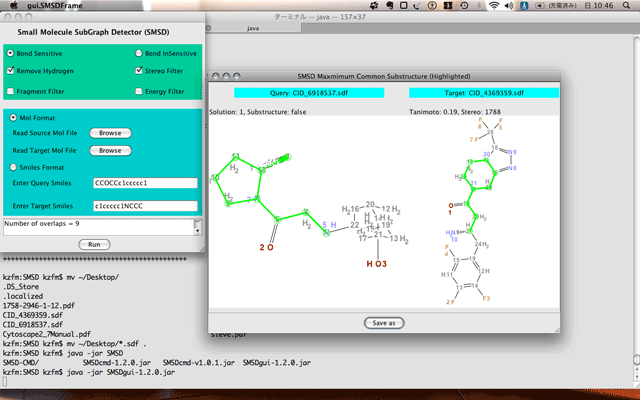
とりあえず、vildagliptinとsitagliptinのMCSをもとめた。
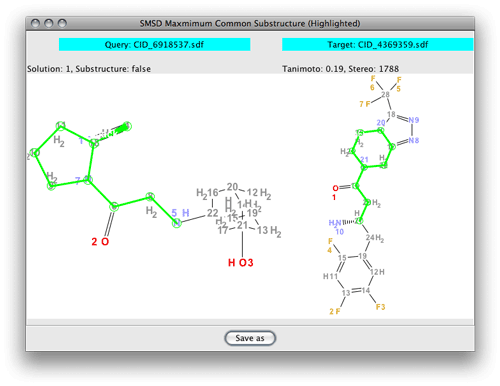
なんで5員環と6員環がマッチしてるんだろうか?あとでペーパーちゃんと読もうっと。
14082010 Python
SWIGでgastonのPythonバインディング作るのがいいのかもしれないけど、SWIG力が足りないので、外部コマンドから使っている。
os.environ['PATH']で探してみたけど、普通はどうやるんだろうか
if 'gaston' not in reduce(lambda a,b: a+b,[os.listdir(d) for d in \
os.environ['PATH'].split(':') if os.path.isdir(d)]):
print "gaston: command not found"
exit()
os.system("gaston %d %s %s > /dev/null 2>&1" % (freq,gasfile,output))
14082010 chemoinformatics cytoscape
ノード間で類似性が最大となるような関係にひとつだけエッジを張るようにしてみた。
#!/usr/bin/env python
# -*- encoding:utf-8 -*-
import pybel
mols = list(pybel.readfile("sdf", "pc_sample.sdf"))
fps = [x.calcfp() for x in mols]
for i in range(len(fps)-1):
max_sim = 0
max_name = ""
for j in range(i+1,len(fps)):
sim = fps[i] | fps[j]
if sim > max_sim:
max_sim = sim
max_name = mols[j].title
print "%s\t%s\t%s\t%2.3f" % (mols[i].title, 'sim', max_name, max_sim)
ついでに類似度属性で色が変わるようにしてみた。
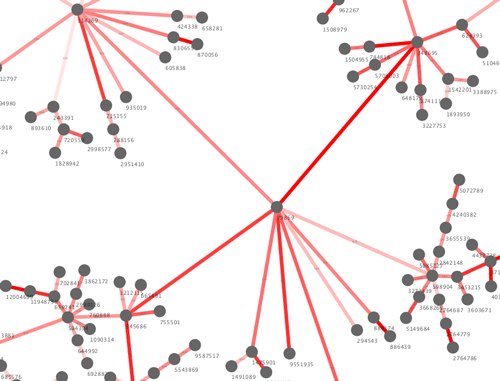
わからん。さっきのエッジに加えて類似性が高い化合物同士のエッジは加えてみた。
for i in range(len(fps)-1):
max_sim = 0
max_name = ""
targets = []
for j in range(i+1,len(fps)):
sim = fps[i] | fps[j]
if sim > max_sim:
max_sim = sim
max_name = mols[j].title
if sim > 0.7:
print "%s\t%s\t%s\t%2.3f" % (mols[i].title, 'sim', mols[j].title, sim)
if len(targets) == 0:
print "%s\t%s\t%s\t%2.3f" % (mols[i].title, 'sim', max_name, max_sim)
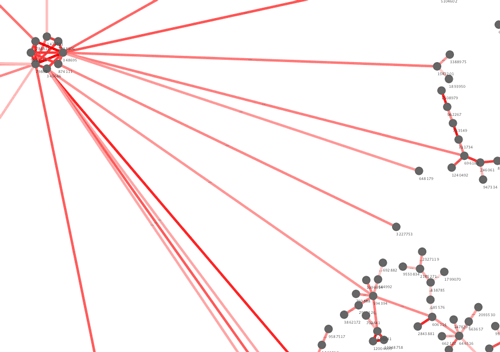
ちょっとまとまりが出てきたけど、あれだなぁ。もとのsdfがそもそもあかんのかなぁ。
で、最小全域木(MST)で描くことも考えたけど化合物間の類似性の最小経路を求めてもなぁ、、、とか思ってヤル気がおきない。類似性でつないだネットワークのMSTの意義ってなんなんだろうなぁと。
なんやろかー、なんやろかー。論文もう一回ちゃんと読んでみようかなぁ。
ブックオフにて。アマゾンの星の多さの割になんというか同意できない内容が多くて、そういう主張を読むという意味ではなかなか興味深かった。労働者は会社に搾取されているっていう前提で、幸せに生きる権利を獲得しなければみたいなスタンスかな。本書からは社員がなんかの目的のために集まって結果できた法人形態が会社っていう立ち位置はさらさら感じさせない。
もちろん正規、非正規にも触れていていて雇用の流動化は悪っていう主張っぽい。
あと、紙切れが挟まってた。栞として便利だったのでそのまま挟んで使った。

ということは著者は社民よりってこと?それとも共産かな、わからん。
理想はまぁその通りだと思うんだけど、それを達成しようとする手段に組合臭を強く感じた。
モチベーション3.0とかハイコンセプトが、自分で未来を切り開こう的な前向きな内容なのに対して、本書は、企業に奪われた「幸せという宝箱」を奪還しようというRPG的な内容かなぁ。まぁ、宝箱の中には「安定雇用」も「正規雇用」も入ってなくてスッカラカンだとおもうんだけどねぇ。