ヒューリスティクスは割と好きだ。スパッととけないものをなんとか現実と折り合いを付けながらよりよい解を見つけるみたいな、なんとも煮え切らない美しさのなさがたまらん。インフォマティクスも似たような香りはする。
特に、「シミュレーションできない学問は学問として未成熟である」という言葉に従うのであれば、創薬研究は(学際領域)と言われている割には未成熟な学問の組み合わせのために発見的な手法の割合が増えすぎるし、精度の高い予測法の誕生もまだまだ先であろう。というわけで、発見的な探索手法は当分有用だし、学際領域故に融合部分での応用が期待されるため、アルゴリズムとしてきちんと押さえておくと色々役に立つ。
メタヒューリスティクスの数理の4章はpythonのコードが載っているので、勉強になる。
- 第4章 応用
- 4.1 グラフ分割問題
- 4.2 最大安定集合問題
- 4.3 グラフ彩色問題
- 4.4 巡回セールスマン問題
- 4.5 2次割当問題
- 4.6 多制約ナップサック問題
- 4.7 数分割問題
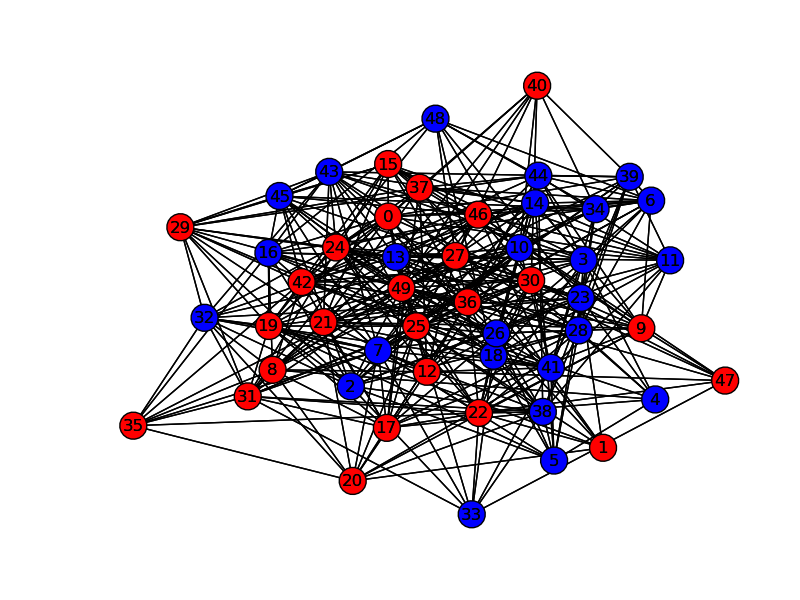
4.1のグラフ分割問題をnetworkxで可視化するように書き換えた。
import math,random
from sys import maxint as Infinity
def rnd_graph_fast(nnodes, prob):
nodes = range(nnodes)
adj = [[] for i in nodes]
i=1;j=1
logp = math.log(1.0 - prob)
while i<nnodes:
logr = math.log(1.0 - random.random())
j += 1+int(logr/logp)
while j>=i and i<nnodes:
j -= i
i += 1
if i<nnodes:
adj[i].append(j)
adj[j].append(i)
return nodes, adj
def construct(nodes):
sol = [0 for i in nodes]
for i in range(len(nodes)/2):
sol[i] = 1
return sol
def evaluate(nodes, adj, sol):
cost = 0
s = [0 for i in nodes]
d = [0 for i in nodes]
for i in nodes:
for j in adj[i]:
if sol[i] == sol[j]:
s[i] += 1
else:
d[i] += 1
for i in nodes:
cost += d[i]
return cost/2, s, d\
def find_move(part, nodes, adj, sol, s, d, tabu, tabulen, iteration):
mindelta = Infinity
istar = None
for i in nodes:
if sol[i] != part:
if tabu[i] <= iteration:
delta = s[i] - d[i]
if delta < mindelta:
mindelta = delta
istar = i
if istar != None:
return istar, mindelta
tabu = [0 for i in nodes]
return find_move(part, nodes , adj, sol, s, d, tabu, tabulen, iteration)
def move(part, nodes, adj, sol, s, d, tabu, tabulen, iteration):
i, delta = find_move(part, nodes, adj, sol, s, d, tabu, tabulen, iteration)
sol[i] = part
tabu[i] = iteration + tabulen
s[i], d[i] = d[i], s[i]
for j in adj[i]:
if sol[j] != part:
s[j] -= 1
d[j] += 1
else:
s[j] += 1
d[j] -= 1
return delta
def tabu_search(nodes, adj, sol, max_iter, tabulen):
cost, s, d = evaluate(nodes, adj, sol)
tabu = [0 for i in nodes]
bestcost = Infinity
for it in range(max_iter):
cost += move(1, nodes, adj, sol, s, d, tabu, tabulen, it)
cost += move(0, nodes, adj, sol, s, d, tabu, tabulen, it)
if cost < bestcost:
bestcost = cost
bestsol = list(sol)
return bestsol, bestcost
if __name__ == "__main__":
import networkx as nx
import matplotlib.pyplot as plt
num_nodes = 50
nodes, adj = rnd_graph_fast(num_nodes,0.3)
G=nx.Graph()
for i in range(num_nodes):
G.add_node(i)
for i in range(num_nodes-1):
for j in adj[i]:
if i < j:
G.add_edge(i,j)
pos=nx.spring_layout(G)
max_iter = 1000
tabulen = 10
sol = construct(nodes)
sol, cost = tabu_search(nodes, adj, sol, max_iter, tabulen)
rnodelist = []
bnodelist = []
for i in range(len(sol)):
if sol[i] == 1:
rnodelist.append(i)
else:
bnodelist.append(i)
nx.draw(G,pos,nodelist=rnodelist,node_color='r')
nx.draw(G,pos,nodelist=bnodelist,node_color='b')
plt.savefig("path.png")
これをちょっと変えてSimulated Annealingにできる。

 社会ネットワーク分析の基礎―社会的関係資本論にむけて
社会ネットワーク分析の基礎―社会的関係資本論にむけて ネットワーク分析 (Rで学ぶデータサイエンス 8)
ネットワーク分析 (Rで学ぶデータサイエンス 8)
 Handbook of Chemoinformatics Algorithms (Chapman & Hall/Crc Mathematical and Computational Biology)
Handbook of Chemoinformatics Algorithms (Chapman & Hall/Crc Mathematical and Computational Biology) Chemoinformatics and Computational Chemical Biology (Methods in Molecular Biology)
Chemoinformatics and Computational Chemical Biology (Methods in Molecular Biology)
 メタヒューリスティクスの数理
メタヒューリスティクスの数理
 複雑な世界、単純な法則 ネットワーク科学の最前線
複雑な世界、単純な法則 ネットワーク科学の最前線 新ネットワーク思考―世界のしくみを読み解く
新ネットワーク思考―世界のしくみを読み解く 複雑ネットワーク入門
複雑ネットワーク入門 最短経路の本
最短経路の本