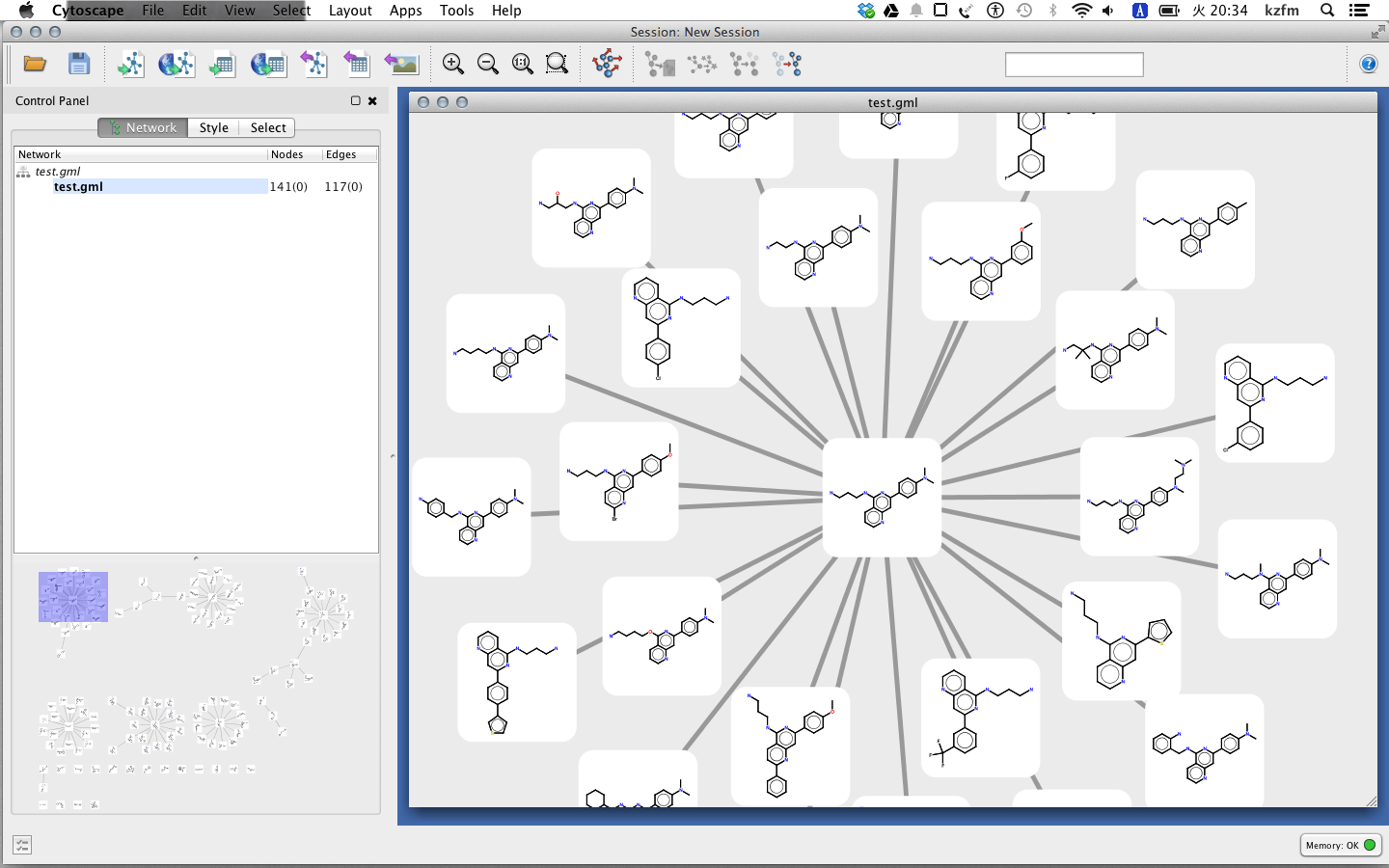
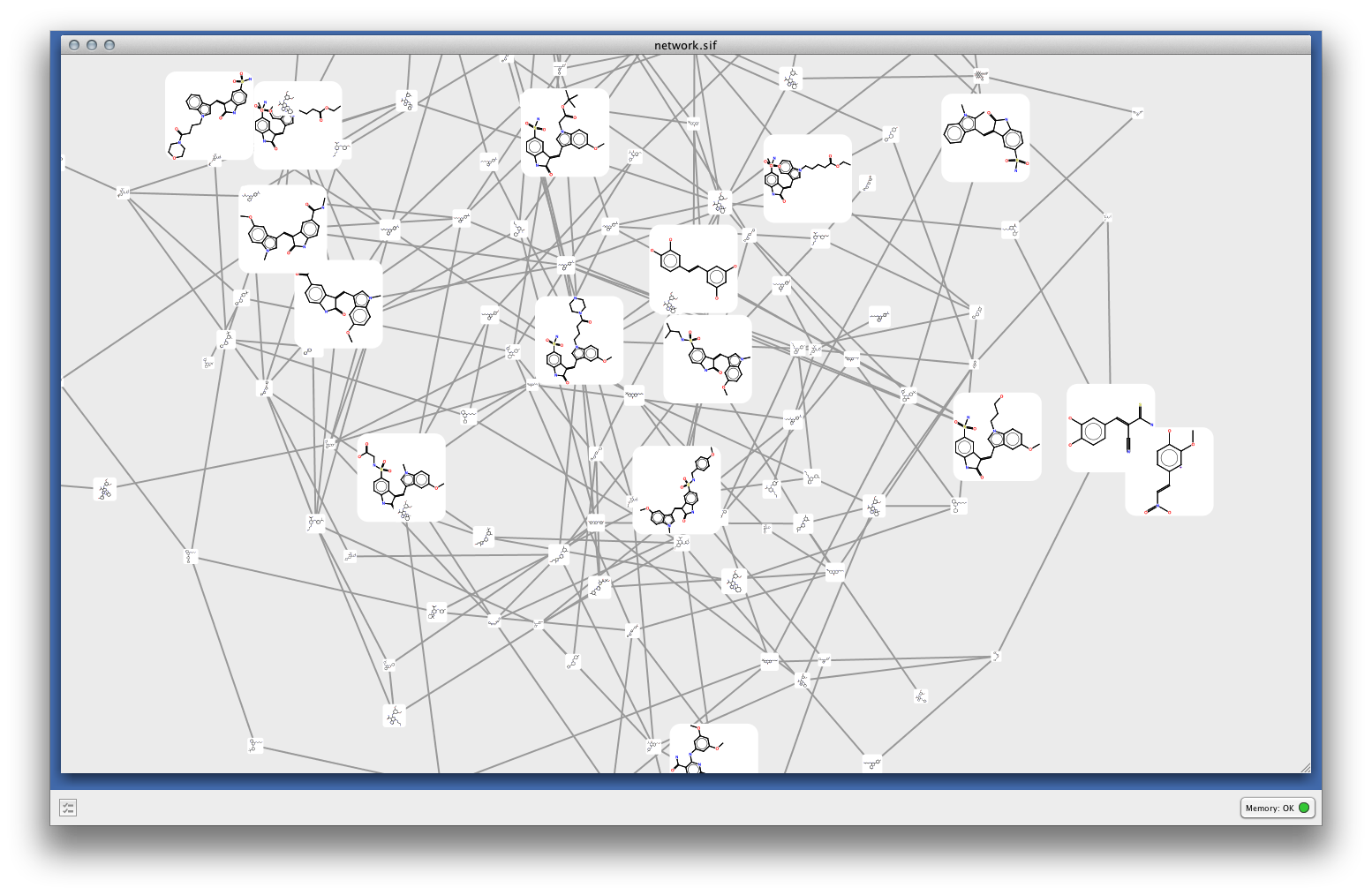
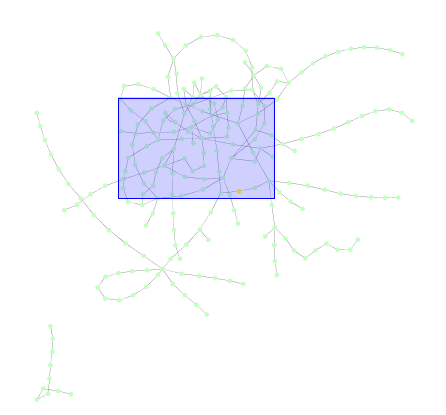
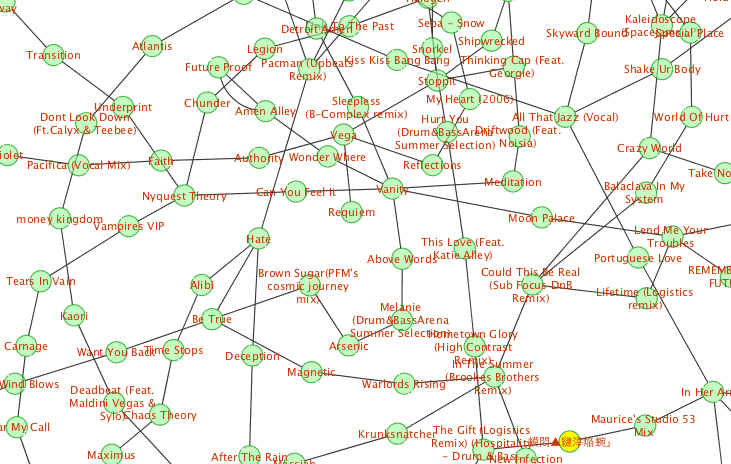
以前、おもむろに思いついたLeadOptimizationのシミュレーターで、networkxはgml出力できてCytoscapeでimportすればいいじゃんということに気づいてヤル気が出た。
ついでにコードもちょっと直して、活性はpIC50とかそういうレンジにしてみて、μMオーダーから上げていくようにした。
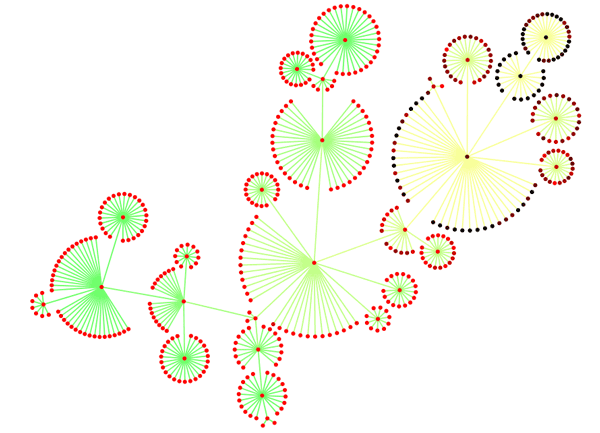
エッジは世代を表していて、黄色から緑にむけてすすんでいく。ノードの色は活性を表していて黒から赤になるにしたがって活性が向上する。最初の方は活性が振れるけど段々向上していって安定するようにしてみた。
リードホッピングとは選択されなかったより良い未来を選択しなおすことであるとするならばそのような希望はLO初期のあたりに存在すると思うのだ。
以下、書いたコード
from random import random
import networkx as nx
class Branch(object):
"""LeadOptimization flow
potency: pIC50 or such
weight : range for activity
"""
count = -1
@classmethod
def inc_count(cls):
cls.count = cls.count + 1
return cls.count
@classmethod
def get_count(cls): return cls.count
def __init__(self,potency,weight):
self.id = Branch.inc_count()
self.potency = potency
self.weight = weight
self.activity = self.potency + self.weight * random()
def make_child(self,num_childs,potency,weight):
return [Branch(self.potency + self.weight * (random()-0.5)*2,self.weight * 0.5) for i in range(num_childs)]
if __name__ == "__main__":
max_comps = 500 # total compounds
initial_branches = 1 # number of lead compounds
lead_potency = 5 # lead activity
generation = 0
G=nx.Graph()
heads = [Branch(lead_potency,2) for i in range(initial_branches)]
map(lambda b: G.add_node(b.id, activity=b.activity),heads)
while Branch.get_count() < max_comps:
new_heads = []
generation += 1
for branch in heads[:int(2+5*random())]:
for new_comp in branch.make_child(int(40*random()),branch.potency,branch.weight):
G.add_node(new_comp.id, activity=new_comp.activity)
G.add_edge(branch.id, new_comp.id, genneration=generation)
if new_comp.activity > 7:
new_heads.append(new_comp)
heads = new_heads
heads.sort(key=lambda x:x.activity)
heads.reverse()
nx.write_gml(G,"test.gml")




 ネットワーク分析 (Rで学ぶデータサイエンス 8)
ネットワーク分析 (Rで学ぶデータサイエンス 8)