14 08 2010 chemoinformatics cytoscape Tweet
ノード間で類似性が最大となるような関係にひとつだけエッジを張るようにしてみた。
#!/usr/bin/env python
# -*- encoding:utf-8 -*-
import pybel
mols = list(pybel.readfile("sdf", "pc_sample.sdf"))
fps = [x.calcfp() for x in mols]
for i in range(len(fps)-1):
max_sim = 0
max_name = ""
for j in range(i+1,len(fps)):
sim = fps[i] | fps[j]
if sim > max_sim:
max_sim = sim
max_name = mols[j].title
print "%s\t%s\t%s\t%2.3f" % (mols[i].title, 'sim', max_name, max_sim)
ついでに類似度属性で色が変わるようにしてみた。
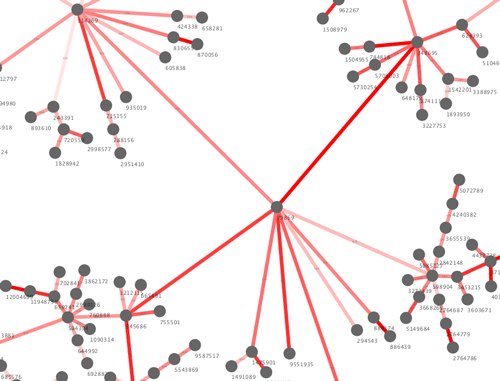
わからん。さっきのエッジに加えて類似性が高い化合物同士のエッジは加えてみた。
for i in range(len(fps)-1):
max_sim = 0
max_name = ""
targets = []
for j in range(i+1,len(fps)):
sim = fps[i] | fps[j]
if sim > max_sim:
max_sim = sim
max_name = mols[j].title
if sim > 0.7:
print "%s\t%s\t%s\t%2.3f" % (mols[i].title, 'sim', mols[j].title, sim)
if len(targets) == 0:
print "%s\t%s\t%s\t%2.3f" % (mols[i].title, 'sim', max_name, max_sim)
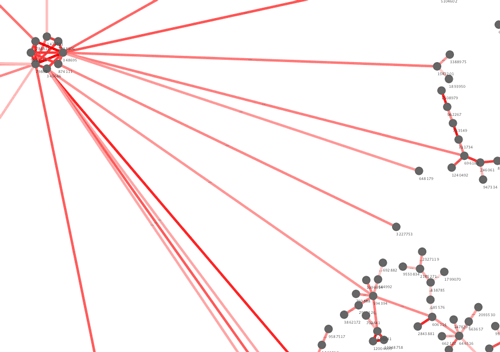
ちょっとまとまりが出てきたけど、あれだなぁ。もとのsdfがそもそもあかんのかなぁ。
で、最小全域木(MST)で描くことも考えたけど化合物間の類似性の最小経路を求めてもなぁ、、、とか思ってヤル気がおきない。類似性でつないだネットワークのMSTの意義ってなんなんだろうなぁと。
なんやろかー、なんやろかー。論文もう一回ちゃんと読んでみようかなぁ。
