sofでグラフ(ネットワーク)を描くためのjavascriptライブラリを見つけたので調べていたら、twitterでCytoscapewebを教えてもらったので、cytoscape使い的にはそっちで色々試してからCytoscapewebで描画できるようにすれば効率的であることに気づき、自分のBlogのタグネットワークを描かせてみることにした。
が、色々つまづいて結局そこまでは至らなかったという。まず、networkxはutf8の属性含んだネットワークをgmlで出力仕様とするとエラーはいた。
しょうがないのでigraphでやるかと思ったらigraphの場合はネットワークを組み立てる場合には結構めんどくさかった(隣接行列から構築する場合には楽だけど)基本的に行列用意しておいて読み込ませるのはいいんだけど、sqlalchemyからオブジェクトをひっぱりつつネットワークを組み立てていくってのはnetworkxのほうがやりやすいようだ。 致命的だったのが、mac osxだとsaveしようとするとBus errorが出てセーブできなかった。
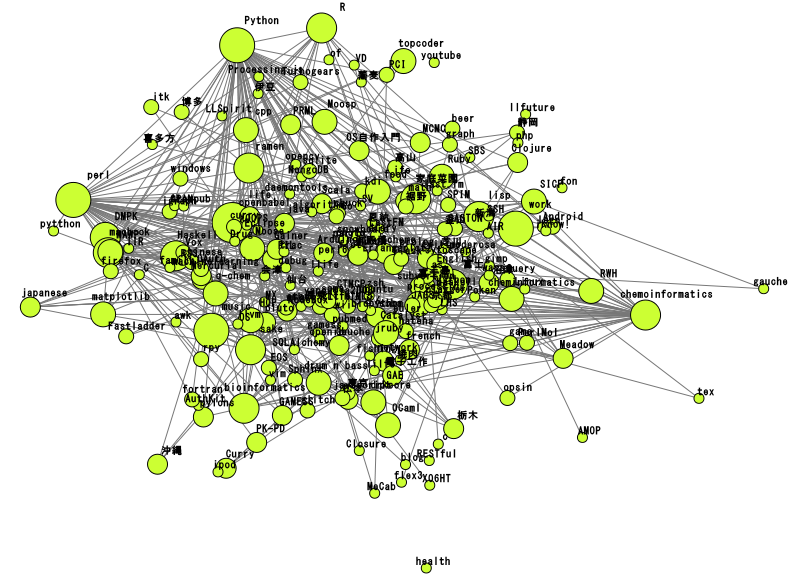
心が折れたので、igraphのプロットのスクリーンショット。
描かせたネットワークは僕のブログのエントリに入ってたタグ名をその頻度でノードの半径が大きくなるようにして、エッジは同じエントリに出現した場合(いわゆる共起)に貼るようにしたもの。
本当はこれをベースにして共起頻度の閾値を決めたり、ノードに最後に現れた日の情報を持たせて古いタグは透明度あげたりしようと思っているので、週末にでもSIFで出力してcytoscapeで綺麗にして適当なフォーマットでエクスポートしてcytoscapewebで見れるようにしてみよう。
ちなみにpythonでigraphをいじるのは下のチュートリアルが良かった。
日本語で書籍になっているのはこれ。
from database import db_session from models import Entry, Tag from math import log import igraph entries = db_session.query(Entry).filter(Entry.publish == 1).order_by(Entry.pubdate.desc()).all() g=igraph.Graph() all_tags = [] for entry in entries: tags = [tag for tag in entry.tags] i = 0 for tag in tags: if tag.name not in all_tags: all_tags.append(tag.name) taghash = dict([(tag,i) for i,tag in enumerate(all_tags)]) tag_count = [0] * len(all_tags) g.add_vertices(len(all_tags)-1) g.vs["name"] = all_tags all_edges = [] for entry in entries: tags = [tag for tag in entry.tags] num_tags = len(tags) if num_tags > 1: for i in range(num_tags-1): itag = tags[i].name tag_count[taghash[itag]] += 1 for j in range(i+1,num_tags): jtag = tags[j].name tag_count[taghash[jtag]] += 1 edge = (taghash[itag],taghash[jtag]) if edge not in all_edges: all_edges.append(edge) else: tag_count[taghash[tags[0].name]] += 1 vsizes = [int(log(tc))*5 + 10 for tc in tag_count] g.add_edges(all_edges) layout = g.layout("kk") g.vs["label"] = g.vs["name"] igraph.plot(g,layout=layout, bbox = (800, 600), edge_color = "#777777",\ vertex_label_size=10, vertex_size=vsizes, vertex_color="#ccff33") #g.write_gml("tag_network.gml")

 ネットワーク分析 (Rで学ぶデータサイエンス 8)
ネットワーク分析 (Rで学ぶデータサイエンス 8) Handbook of Chemoinformatics Algorithms (Chapman & Hall/Crc Mathematical and Computational Biology)
Handbook of Chemoinformatics Algorithms (Chapman & Hall/Crc Mathematical and Computational Biology) Chemoinformatics and Computational Chemical Biology (Methods in Molecular Biology)
Chemoinformatics and Computational Chemical Biology (Methods in Molecular Biology)