24102006 perl
知らんかった。というかAnnoCPAN自体ちゃんとわかっていなかったので読んでみた。
なかなか面白そうなので、アカウントもとってみた。
24102006 perl
Chemistry::File::SMARTS Version 0.22
implicit,explicit
implicit:明示的に指定されていないbondとか(水素)原子も含んだ完全な形で。
smiles記法では(明らかに省略できる)水素原子は省略するのでexplicit,implicitという区分けがある。MOLもそうだけど。
などを参照するとイメージはわかるはず。
パターンマッチの確認は以下のコードでおこなった。
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 | #!/usr/bin/perl use warnings; use strict; use Chemistry::File::SMARTS; use Chemistry::File::SMILES; use Chemistry::Ring 'aromatize_mol'; my ($SMARTS,$SMILES) = @ARGV; print "SMARTS: $SMARTS\n"; print "SMILES: $SMILES\n"; my $pattern = Chemistry::Pattern->parse($SMARTS, format => 'smarts'); my $react = Chemistry::Mol->parse($SMILES, format => 'smiles'); aromatize_mol($react); my $count = 0; while ($pattern->match($react)){ $count++; } print $count,"\n"; |
*
全ての原子にマッチ
a,A
Aromatic(a) or Aliphatic(A)。
PerlMolではChemistry::Ringでaromatize_molしとかないと認識しない。
D<n>,X<n>,x<n>
結合原子数(n)を有するAtomにマッチ。明示的(D)か、内在的(X)か。つまり、Dは明示的にHとの結合が指定されていない限りこれをカウントしない。xはリングのコネクティビティだが、PerlMolではサポートされてないっぽい。
H<n>,h<n>
明示的なHをカウントする(H)か内在的なものも含めるか(h)
R<n>,r<n>
n原子からなるSmallest Set of Smallest Rings (SSSR)のメンバーにマッチ(R)。PerlMolだとRかどうかだけしか判断していないので<n>は関係ないかも。リングサイズが最小のSSSRのメンバーにマッチ(r)。芳香環かそうでないかは判断せず、n員環のメンバーであればマッチ
こんな感じ
$ ./smartstest [r6] 'C1CCCCC1CCC' # 6 $ ./smartstest [r6] 'c1ccccc1CCC' # 6 $ ./smartstest [r6] 'c1cc1CCC' # 0 $ ./smartstest [r3] 'c1cc1CCC' # 3
v<n> バレンス。いわゆる手を伸ばせる数(Nは3で、Cは4)
<n>,+<n>,#n チャージの有無と原子番号
@,@@,@<c><n>,@<c><n>? キラルはPerlMolではサポートされてない。
下の例
$ ./smartstest "c:c" 'c1ccccc1' SMARTS: c:c SMILES: c1ccccc1 6 $ ./smartstest "c=c" 'c1ccccc1' SMARTS: c=c SMILES: c1ccccc1 3 $ ./smartstest "c-c" 'c1ccccc1' SMARTS: c-c SMILES: c1ccccc1 0
~,@ ワイルドカード(~)、リング結合全て(@)
/,,/?,? PerlMolでサポートされてるかどうか調べてない(とりあえず使わないので後回し)
否定(!),論理積(&,; ただし、&は最初に評価される),論理和(,)
PerlMolではサポートされていない
こういうのをblogに突っ込むのは無理がある。素直にWiki+Markdown入れようと思ったヨ。
smilesを描画するwebのインターフェースが欲しくて、色々作ってみた
必要なもの
Chemistry::OpenBabel( need for iupac2png)
http://depth-first.com/articles/2006/10/17/from-iupac-nomenclature-to-2-d-structures-with-opsin
ここら辺を参考に書いた。できたものはCDK-Util-0.01.tar.gzにおいておいた。
が可能。IUPAC2PNGも書いたのだけどmake testで豪快にこけるので無視してインストールしてください。podの細かいとこも気にしないように。Iupac2Pngはcdkでcmlをmolに変換するあたりがわからなくてあきらめたけど、そこのコードの部分だけ手直しすれば動くと思う。
っていうか、iupac2pngはopenbabelのモジュール使えばいいじゃんって気付いた途端もういいやってなってしまった。書式コンバートはopenbabelのほうがわかりやすい。ちょっとコード量は増えるけど、こんな感じ。
use strict; use warnings; use CDK::Smi2Png; use CDK::Iupac2Cml; use Chemistry::OpenBabel; my $iupac_name = shift; my $ic = CDK::Iupac2Cml->new(); my $cml = $ic->convert($iupac_name); my $obMol = new Chemistry::OpenBabel::OBMol; my $obConversion = new Chemistry::OpenBabel::OBConversion; $obConversion->SetInAndOutFormats("cml", "smi"); $obConversion->ReadString($obMol, $cml); my $smi = $obConversion->WriteString($obMol) or die "$! not converted"; $smi = (split(/\t/, $smi))[0]; my $smic = CDK::Smi2Png->new(); $smic->writePNG($smi,200,200,'/home/kzfm/test.png');
で実行する。Imatinibくらいは認識するようだ。
./iupac2png.pl "4-[(4-methylpiperazin-1-yl)methyl] \ -N-[4-methyl-3-[(4-pyridin-3-ylpyrimidin-2-yl)amino] \ -phenyl]-benzamide"
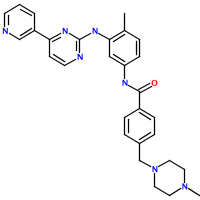
あとはcatalystとsmi2pngを組み合わせて、chemichoのwebインターフェースに使ったり、iupac2pngでcgiでもつくってbookmarkletでポストできるようにしとけばpubmedとかのIUPAC名をいつでもひける。辞書サーチのプラグインで右クリック呼び出しでもいいけど。
いまいち、Class::DBIとDBIx::Classの違いがわかっていないのだけど、Catalystで作りたいものがあったので Perl/DBICなどみながら、いろいろさわってみてる。
Class::DBI inspired ORM designed to be resultset-oriented rather than table-oriented - so native support for views, aggregates (GROUP BY etc.), better automatic JOIN-ing and the ::Sweet search features done properly.
Class::DBIがテーブル指向なのに対して、DBIx::Classはresultset指向らしい。確かにCDBIだとページの処理用にプラグインがあったりするけどDBIx::Classだとさくさく書けた。
もうちょいドキュメント 読む必要があるけど、なかなか楽しげ。
ちなみに、下の二つは同じらしい。今日書いててちょっと悩んだのでメモ
$c->model('DBIC')->resultset('Items')
DBIx::Class::Manual::Glossary - Clarification of terms used. - search.cpan.org
ResultSetThis is an object representing a set of data. It can either be an entire table, or the results of a query. The actual data is not held in the ResultSet, it is only a description of how to fetch the data.
ResultSetとはデータセットを表すオブジェクトで、テーブル全体であったり、クエリの結果だったりする。
あーそういえば、BXRってのがあったなというのを思い出したので、Net::MovableTypeで投稿することを思いついた。
my $mt = new Net::MovableType('http://localhost/blosxom-xmlrpc.cgi/RPC2'); $mt->username('kzfm'); $mt->password('okbkj'); $mt->blogId(1); $entry = { title => "Hello World from Net::MovableType", description => "おーけーおーけーOK牧場" }; $mt->newPost($entry);

おー、かなりOK牧場だ。
ちなみにpackage blosxomのnewPostをちょっといじって常にランダムなファイル名を付与するようにした。ファイル名考えなくていいってだけでも精神的にはかなりラク。
# if ( defined( $struct->{'postid'} ) ) { # $filename = BXR::getFilenameFromPostId( $struct->{'postid'} ); # } # else { # $filename = lc( $struct->{'title'} ); # $filename =~ s/\W+/_/g; # $filename =~ s/_+$//; # $filename = BXR::getRandomFilename() unless $filename =~ m/[a-z]/;
これで、Plagger::Plugin::Publish::MTを使ってChangelog2blosxomができるはず。ここまでやれれば、僕はchangelogだけに集中すればよくて、それ以降の処理はplaggerがよきにはからってくれるのでかなり調子いいかも。あとは学会とかセミナーの参加ログもchangelogでとってるのでms-wordにpublishしたいなぁ。
12102006 perl
perlのテストのやり方って、みんなどうやって覚えるんだろうか?と疑問に思ってる(今でも)。そっち系の会社だと教育みたいなのあるんだろうなとか?
でも、僕の場合は周りにプログラミングするヒトがいなかったので、テスト技法っていまいちよくわかってなかった。続・初めてのperlにもちょろっとしか書いてないし、あれじゃぁ、実際のテストコード書けるようにならんよなぁとか思ってた。そういうわけで、なかなかプリントデバッグから先に進まない。しかもよくわからんので、モジュールのソースは読むけど.tは読まんみたいな。
というわけで、テストは覚えたいけど、何を勉強すればよいのかわからんという状態だったが、最近、Perl Testingが色々なところで薦められていたのでこれはいかねばと、買って読んでみた。
 Perl Testing: A Developer's Notebook (Developers Notebook)
Perl Testing: A Developer's Notebook (Developers Notebook)かなりわかりやすくて、さくさく読めた。事例から学ぶノートブックというスタイルで、最初のほうは基本的なことから始まり、徐々にデータベースのテストとかwebアプリのテストまで押さえてある。読み終えたら、あとはドンドコ書いて、cpanの.tをドンドコ読むと。
というわけで、最近は.tのファイルも読んでて楽しい。もちろんデバッグも楽になった。
11102006 chemoinformatics perl plagger planet
planet chemoinformaticsのRSS Auto Discoveryが微妙におかしい(/が抜けてた)ので修正。これで、livedoor readerとかbloglinesに登録できる。ま、直接rssを放り込んでも良いんだけど。僕はwebでほとんどみないので、デフォルトのテンプレートをいじって綺麗にしたいという欲求が沸いてこない(今のとこ)。だからRSSリーダーで読んだほうが良いと思うヨ。ちなみに、一日3回更新するように設定してます。
あと、docking.orgも追加した。rssが日付と本文を含まないので、Filter::EntryFullTextを書いてみた。
author: kzfm handle: http://docking.org/.*?article\.pl\? extract: (<TABLE WIDTH="100%" BORDER="0" CELLPADDING="5" CELLSPACING="0"> <TR><TD BGCOLOR="#FFFFFF">.*? </TD></TR></TABLE>) .*?NAME="returnto" VALUE="//docking.org/article.pl\?sid=(\d\d/\d\d/\d\d)/\d+"> extract_capture: body date extract_date_format: %y/%m/%d
なかなか快適になってきた。
10102006 perl
Class::DBI::Loader::Relationshipが便利そうなので、ドキュメント眺めてたら、
Class::DBI::Loader::Relationship - Easier relationship specification in CDBI::L - search.cpan.org
This module acts as a mix-in, adding the relationship method to Class::DBI::Loader.
とかいう記述があったので、何してるんだろかとソース眺め。
結局Class::DBI::Loader::Relationship自体は何もしてナイっぽくて、Class::DBI::Loader::Genericの名前空間でrelationshipメソッドが記述されてた。
うーんpackageって同じ名前空間を複数回宣言していいのか?と不思議だったので、試したのが以下のコード。
package A; sub a1_test { print "a_1\n";} sub a2_test { print "a_2\n";} 1; package B; sub b1_test { print "b_1\n";} sub b2_test { print "b_2\n";} 1; package C; sub c1_test { print "c_1\n";} sub c2_test { print "c_2\n";} 1; package A; sub a3_test { print "a_3\n";} 1; package Main; A::a1_test(); B::b1_test(); C::c2_test(); A::a3_test();
で、実行。果たしてa3_testは呼び出されるんだろうか?
$ perl packtest.pl a_1 b_1 c_2 a_3
うーん、これでもいいのね。
ただmix-inに関してはどっかから借りてくるのをmix-inって呼ぶのかナァと漠然と感じていたので、こういうやり方をmix-inと呼ぶのが, アイスクリームにアイスクリームを混ぜ込んでるみたいで、ちょっと不思議な感じがするんだよなぁ。
06102006 perl
どっかで、
1 while (unlink $file)
っていう記述を見つけたので、何これ?と調べてみたら定番だった。
つまり、「~が失敗するまで~を続ける」という定番の 書き方ですね。
02102006 perl
プロキシーサーバーの設定は
$search->http_proxy(['http','ftp'] => 'http://proxy:port');
で。$search->env_proxyはなぜかだめ。
ここらへんのモジュールを使って、P450 Tableから、CYPの基質とか阻害剤の情報取ってきて、DrugBankの構造情報とつなげてみた。
いきなり、P450 TableはDOMれず、躓く。しょうがないので、正規表現使って化合物名とinhibitorとかのクラス分類を抽出した。あとは、WWW::Search::DrugBank 使って、ほしいデータ(mol)をサーチしてつなげれば完了とか思ったんだけど、WWW::Search::DrugBankってIDでしか検索できない。うーん。
結局、webからサーチかます(これも色々ヒットしてきて精度悪くないか?)サブルーチン用意して、IDゲットしてからWWW::Search::DrugBank使って欲しいデータ取ってきた。