28112013 perl
Mishima.pmが開催されるらしい。
でも平日の夜…
28112013 perl
30062013 perl drum'n'bass Python
radrがTwitter API1.1に対応したらしいが、先週Traktor-SongNameServerというものを見つけたのでこちらに乗り換えた。
traits => [qw/OAuth API::RESTv1_1/],
って変更だけで動くし。
ログとっているのでperlからsqliteにアクセスするかTraktor-SongNameServerをPythonで書きなおしてリアルタイムで収集するようにしたい(今はプレイリストをXMLにエクスポートしてそれを読んでる)
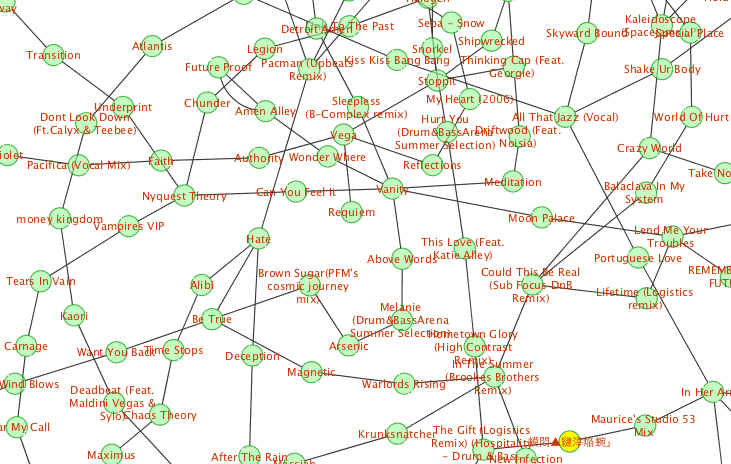
ついでに、曲の雰囲気をコメントに入れているので、配色イメージを参考にしてノードに色つけすればいいか。そうすれば選曲をもっと楽しめるはず。
Darkling Sky / Barefoot Electric Smile / Dom & Roland Inspired By Insane / Fade Cant You See / Optiv & CZA Right Here / Ulterior Motive Dead Hollywood / Royalston Whatever (Mefjus Remix) / Optiv & BTK Circles / Enei featuring Sam Wills The Chant / Mikal Boshoven / Nymfo & June Miller Marka (feat. Strategy) / Dub Phizix & Skeptical Run / Spinline We Are One (NuLogic Remix) / Logistics Rich Kids / Hamilton Boiler Room / Ben Fawce & P Fine We Become One (feat. Foreign Beggars & Craze) / Calyx & TeeBee Posers / Breakage To Fly / Eastcolors & Enei Ice Block / James Marvel & Shimon This City (12 Mix) / LSB feat Sian Sanderson Liberation / Enei & Emperor Midnight Nation / Optiv Chubrub / Various Artists Temper / Ed Rush & Optical Roxy / Nymfo & State Of Mind Sleepless (B-Complex remix) / New Zealand Shapeshifter Hide Your Tears Because We Are In Heaven / Unquote
17022012 chemoinformatics perl
Perlでコードを書いていてCytoscape用にファイルを出力したかったんだがGraph::GMLは読み込み専用だし、Graph::XGMMLはCytoscapeじゃ読み込めないし、、、
Graph::XGMMLのソースコードを読んでみたら、XML::Writerを使えばいいみたいなので書いた。
sub write_xgmml { open my $output, '>', 'output.xgmml'; my $xml = XML::Writer->new(OUTPUT=>$output); $xml->xmlDecl('UTF-8'); $xml->startTag('graph', directed=>"1", label=>"Sample1", 'xmlns:dc'=>"http://purl.org/dc/elements/1.1/", 'xmlns:xlink'=>"http://www.w3.org/1999/xlink", 'xmlns:rdf'=>"http://www.w3.org/1999/02/22-rdf-syntax-ns#", 'xmlns:cy'=>"http://www.cytoscape.org", 'xmlns'=>"http://www.cs.rpi.edu/XGMML" ); my $i = 1; my %dict; #add node foreach my $key ( keys %$ids ){ $dict{$key} = $i; $xml->startTag('node', id => $i, label => $key, ); $xml->emptyTag('att', type => 'string', name => 'canonicalName', value => $key ); $xml->emptyTag('att', type => 'real', name => 'property', value => $ids->{$key}->{'property'} ? $ids->{$key}->{'property'} : 0.0 ); $xml->endTag('node'); $i++; } #add edge for my $e (@$spt) { $xml->startTag('edge', label => "$e->[0] to $e->[1]", source => $dict{$e->[0]}, target => $dict{$e->[1]} ); $xml->emptyTag('att', type=>'string', name=>'canonicalName', value=> "$e->[0] to $e->[1]" ); $xml->endTag('edge'); } $xml->endTag('graph'); $xml->end; close($output); }
基本的にstartTagとendTagで包んでemptyTagで属性を入れてくだけですね。あとCytoscapeのIDって整数じゃないとダメだったような気がするので、そうなるように書いたんだけど実際どうだったかは忘れた。
やたらとこけるJSONを出力する商用のツールを追いかけていたら、浮動小数点の表記に問題があった。1.e-2っていうのはJSON的にはmalformed numberなんだろうか?
use strict; use warnings; use JSON; print decode_json('{"num": 1.e-2}')->{'num'};
実行
$ perl jsontest.pl malformed number (no digits after decimal point), at character offset 10 (before "e-2}") at jsontest.pl line 5.
>>> import json >>> json.loads('{"num":1.0e-2}') {u'num': 0.01} >>> json.loads('{"num":1e-2}') {u'num': 0.01} >>> json.loads('{"num":1.e-2}') Traceback (most recent call last): File "<stdin>", line 1, in <module> File "/usr/local/Cellar/python/2.7.2/lib/python2.7/json/__init__.py", line 326, in loads return _default_decoder.decode(s) File "/usr/local/Cellar/python/2.7.2/lib/python2.7/json/decoder.py", line 366, in decode obj, end = self.raw_decode(s, idx=_w(s, 0).end()) File "/usr/local/Cellar/python/2.7.2/lib/python2.7/json/decoder.py", line 382, in raw_decode obj, end = self.scan_once(s, idx) ValueError: Expecting , delimiter: line 1 column 8 (char 8)
12022012 perl
インフラまわりをやらせてもらえるようになったので、古いCatalystで動いているシステムを一新したくて何に置き換えようかと悩んでいる
これをみるとDancerがいいような気がしてくるんだけど(いつもはFlask使っているので)。
24012012 perl javascript
アルゴリズムの勉強のしかたに載っていて、java度の低い自分には丁度よいかもと思い即買いしたのだけど、自分にはちょっと基礎的すぎた(どういうライブラリがあるかとリングバッファの実装が役に立ったけど)。
アルゴリズムイントロダクションを買うべきだったな。
ところでperlでアルゴリズムを学ぶなら迷わずMAWPを選択するべきでしょう。
日本語版がでてもおかしくないくらいの良書だと思う。
04062011 perl javascript
第1回 静岡ITPro勉強会 インフラ部で発表してきた。デモが動かなかったのは、名前がダサいという理由で電車の中でhtmlのファイル名を変えたせいでした、アホ過ぎる。
ま、こんな感じのファイルを用意します。AjaxでATNDのAPIにアクセスして今日の参加者を表示するっていうやつです。
<!DOCTYPE html> <html> <head> <title>ajaxtest</title> <script type="text/javascript" src="jquery-1.6.1.min.js"></script> </head> <body> <button id=ajax name="atnd">クリックするとユーザー一覧を表示します</button> <div class=users></div> <script> $("button#ajax").click(function(){ $.getJSON('http://api.atnd.org/events/users/?event_id=16076&format=jsonp&callback=?', function(data){ var items = []; $.each(data.events[0].users, function() { items.push('<li>' + this.nickname + '</li>'); }); $('<ul/>', { 'class': 'userlist', html: items.join('') }).appendTo('div.users'); }); }); </script> </body> </html>
これをスクレイピングするにはWWW::Mechanize::Firefoxを使って
use WWW::Mechanize::Firefox; use Web::Query; use Encode; my $mech = WWW::Mechanize::Firefox->new(); $mech->get('http://localhost:8000/'); $mech->click({ xpath => '//button[@name="atnd"]', synchronize => 0 }); sleep(2); #print $mech->content; my $q = Web::Query->new_from_html($mech->content); $q->find('li')->each(sub { my $i = shift; printf "(%d) %s\n", $i+1, encode('utf-8', $_->text); });
実行するとこんな風に出力されるはずです。
(1) secondarykey (2) ando_ando_ando (3) となか (4) yukio.47 (5) Kaz110 (6) tatsuya.ueda (7) taji_314159265 (8) kzfm (9) non
凡ミスでここまでは行かなかった。カナシス
14042011 perl
これを読めばPerlグルメまちがいなしですね。Perlの経験がある程度あれば、モジュールを組み合わせて素敵なコードを紡ぎ出すという新たな楽しみを見いだせるようになるでしょう。
個人的には表紙がCPANの世界観をとてもよく表現していて素敵だと思った。
一通り読んでみて、興味深かったモジュール群。
最近コマンドラインのツールを作ることが多いのでそういう作業を楽にするようなモジュールに偏っているかも。
モジュールガイドなので継続的にアップデートされると嬉しいですが、ここらへんは書籍の限界ですかね。
perl使いは是非手元に置いておくといいと思います。
せっかくなのでこの前書いたファイル監視スクリプトをFilesys::Notify::Simpleを使って書きなおしてみた。
use Filesys::Notify::Simple; my $watcher = Filesys::Notify::Simple->new(['.']); while(1) { $watcher->wait(sub { for my $event (@_) { next unless $event->{path} =~ /\/([\w\d]+)\.rst$/; print "### ", $event->{path}, " ###\n"; system("make html"); } }); }
余談だが、Mac::FSEventsを内部的に使ったらmacbookではクラッシュしたのでpm-uninstallでアンインストールした。
簡単にuninstallできたり、更新できたりとcpanmのお手軽さは素晴らしいですね。
pdbgetというpdb-idを引数にとって構造データをまとめてダウンロードするコマンド使っていたんだけど、ちょっと前のサーバリプレースでPDBのミラリング環境が失われたので使えなくなってた。こういうの致命的だよなとは思うが今時はネットワークが太いので別にミラリングする必要性はあまりないのかな?でも遅いよなぁ。
とりあえず、自分用に。プロキシ使いたいのでFancyURLopener
import urllib import sys pdblist = sys.argv[1:] proxies = {'http': 'http://[hostname]:[port]/'} opener = urllib.FancyURLopener(proxies) for pdbid in pdblist: f = opener.open('http://www.rcsb.org/pdb/files/%s.pdb.gz' % pdbid.upper()) data = f.read() with open('%s.pdb.gz' % pdbid.upper(),'wb') as wf: wf.write(data)
ちなみに初代はperlで書いてあった。これいつ書いたんだろう?多分7,8年よりは前だと思うんだよなぁ。
use strict; use Net::FTP; die "usage:$0 [pdb_id] ...\n" unless @ARGV; my $ftp = Net::FTP->new("[hostname]", Debug => 0) or die "Cannot connect to [hostname]: $@"; $ftp->login("pdb",'pdb') or die "Cannot login ", $ftp->message; $ftp->binary(); for my $pdbid (@ARGV){ $pdbid =~ tr/[A-Z]/[a-z]/; my $pdbdir = substr($pdbid,1,2); my $pdbfile = "pdb" . $pdbid . ".ent.gz"; $ftp->cwd($pdbdir) or warn "Cannot change PDB directory ", $ftp->message; $ftp->get($pdbfile) or warn "$pdbfile : ", $ftp->message; $ftp->cwd('..') or warn "Cannot change PDB directory ", $ftp->message; } $ftp->quit;
 Bioinformatics Programming Using Python
Bioinformatics Programming Using Python