smilesを描画するwebのインターフェースが欲しくて、色々作ってみた
必要なもの
- Java
- Structure-CDK (include cdk)
- opsin-big-0.1.0
- Perl
- Inline::Java
-
Chemistry::OpenBabel( need for iupac2png)
-
http://depth-first.com/articles/2006/10/17/from-iupac-nomenclature-to-2-d-structures-with-opsin
- http://depth-first.com/articles/2006/08/28/drawing-2-d-structures-with-structure-cdk
- http://sourceforge.net/projects/structure
ここら辺を参考に書いた。できたものはCDK-Util-0.01.tar.gzにおいておいた。
- smi2mol
- smi2png
- IUPAC2CML
が可能。IUPAC2PNGも書いたのだけどmake testで豪快にこけるので無視してインストールしてください。podの細かいとこも気にしないように。Iupac2Pngはcdkでcmlをmolに変換するあたりがわからなくてあきらめたけど、そこのコードの部分だけ手直しすれば動くと思う。
っていうか、iupac2pngはopenbabelのモジュール使えばいいじゃんって気付いた途端もういいやってなってしまった。書式コンバートはopenbabelのほうがわかりやすい。ちょっとコード量は増えるけど、こんな感じ。
use strict; use warnings; use CDK::Smi2Png; use CDK::Iupac2Cml; use Chemistry::OpenBabel; my $iupac_name = shift; my $ic = CDK::Iupac2Cml->new(); my $cml = $ic->convert($iupac_name); my $obMol = new Chemistry::OpenBabel::OBMol; my $obConversion = new Chemistry::OpenBabel::OBConversion; $obConversion->SetInAndOutFormats("cml", "smi"); $obConversion->ReadString($obMol, $cml); my $smi = $obConversion->WriteString($obMol) or die "$! not converted"; $smi = (split(/\t/, $smi))[0]; my $smic = CDK::Smi2Png->new(); $smic->writePNG($smi,200,200,'/home/kzfm/test.png');
で実行する。Imatinibくらいは認識するようだ。
./iupac2png.pl "4-[(4-methylpiperazin-1-yl)methyl] \ -N-[4-methyl-3-[(4-pyridin-3-ylpyrimidin-2-yl)amino] \ -phenyl]-benzamide"
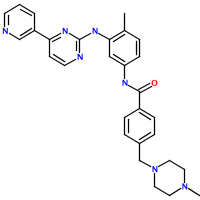
あとはcatalystとsmi2pngを組み合わせて、chemichoのwebインターフェースに使ったり、iupac2pngでcgiでもつくってbookmarkletでポストできるようにしとけばpubmedとかのIUPAC名をいつでもひける。辞書サーチのプラグインで右クリック呼び出しでもいいけど。
