10 08 2014 chemoinformatics cytoscape Tweet
Chemviz2を使い始めたのでメモ
インストール
- Cytoscapeのサイトから3.xの最新バージョンをダウンロードしてインストール
- Apps -> App ManagerからChemviz2を探してインストール
使ってみる
pychembldbを使ってノード用の属性ファイルとnetworkファイルを作った。ネットワークはとりあえずランダムにつないでみた。
Cytoscapeを起動して"network.sif"をインポート。続いてテーブルファイルとして"node.csv"をimport プライマリキーはsmilesにする。
構造描画の設定はApps -> Chemoinformatics Tools -> SettingsでSmiles Attributesをnode.shared.nameを選んでおく。 構造描画するときには右クリックしてオプションダイアログのApps -> Cheminformatics Toolsでpaint allかselectを選ぶ。
サイズをpIC50にして、オーガニックレイアウトで表示してみた。なかなかいい感じ
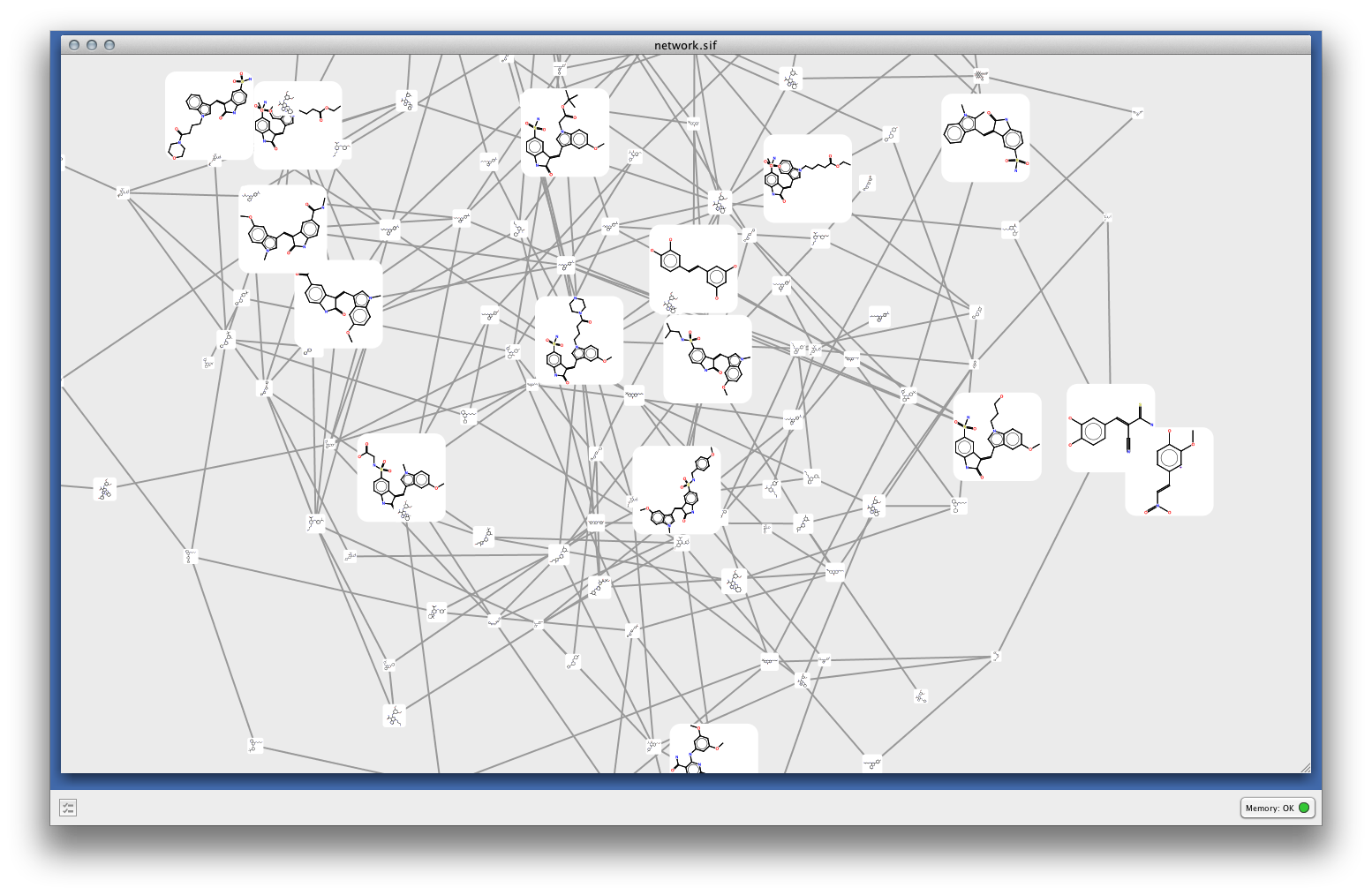
よく分からなかったところ
- nameとshared nameの違いがよく分からなくてドキュメントあたってみたけど結局良くわからなかった
- Chemviz2でsmilesをshared nameにしなきゃいけなくてsifファイルをsmilesで作ったのがダサかった
- (sifのidはchembl_idとかにしたかったけど)
コード
from pychembldb import * from copy import copy from random import shuffle, random from math import log10 #Inhibition of recombinant Syk #Bioorg. Med. Chem. Lett. (2009) 19:1944-1949 assay = chembldb.query(Assay).filter_by(chembl_id="CHEMBL1022010").one() with open("node.csv","w") as f: f.write('"ID","pIC50","ALOGP","MWT","SMILES"\n') for act in assay.activities: if act.standard_relation == "=": f.write('"{}",{},{},{},{}\n'.format(act.compound.molecule.chembl_id, 9 - log10(act.standard_value), act.compound.molecule.property.alogp, act.compound.molecule.property.mw_freebase, act.compound.molecule.structure.canonical_smiles)) with open("network.sif", "w") as f: smiles_list = [ act.compound.molecule.structure.canonical_smiles for act in assay.activities if act.standard_relation == "="] for i in range(len(smiles_list)): tl = copy(smiles_list) tl[i:i+1] = [] shuffle(tl) f.write('{} randomnetwork {}\n'.format(smiles_list[i], " ".join(tl[:int(random() * 5)])))
