04 08 2012 chemoinformatics cytoscape Tweet
複数のパラメータが相互作用しあっている状態で最適なパラメーターセットを探索する際に、あまり精度の高いフィードバックが得られないと最適解の近傍の密度が高くなり、さらに準最適解があちこちに散らばるようなことがおこる。
それはPCAとかICAで空間を適当にとってあげることが出来て、特に第一主成分と第二主成分を平面にとってプロットしてやると、散布図として表現できる。
さてこれを、グラフにしてやればCytoscapeで扱えて便利だろうと思ったことがあったのでやり方を考えてみた。
アルゴリズム
- 任意の点から非類似度Tの距離にある点の数をすべての点について数える
- 最も点を含むものが多い点をrootとし、含まれる点との間で親子関係をつくる
- 2の点を除いて1を行い新たな親子を決める。その際に親から非類似度T内にある既存の子のうち一番近いものを結ぶ(階層ができる)
- これをすべての点がなくなるまで繰り返す。
例
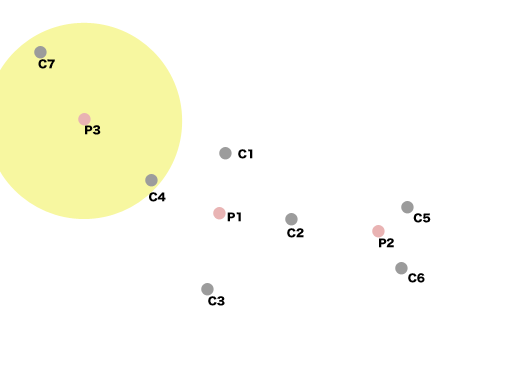
以下の様な散布図を考える。

総当りで近傍の数をかぞえると、次の5つの点が含まれる空間が一番密度が高いことが分かる。

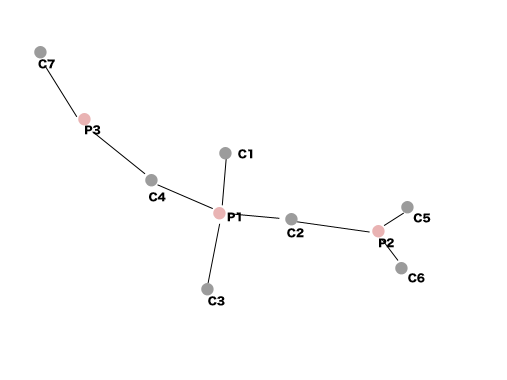
中心と含まれる点に対して親子関係をつくる(P1, C1-C4)。
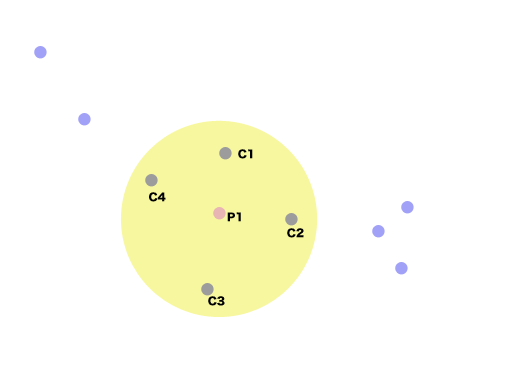
これらを除いて、近傍の数をかぞえると次に密度が高いのが右側で3つの点を含んでいる。
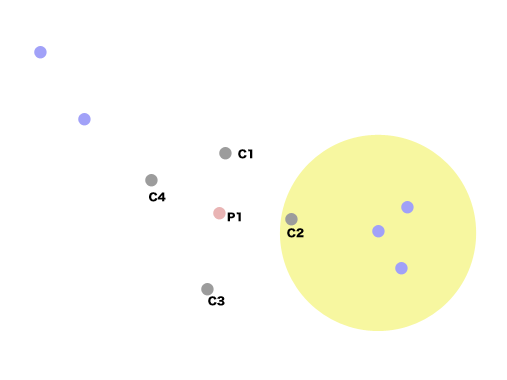
これらに関しても先ほどと同様に親子関係をつくっておくが、新しくできた親に関しては既存の子とつなげられないかをチェックする。
するとC2 -> P2をつなげるのが良いことがわかる。
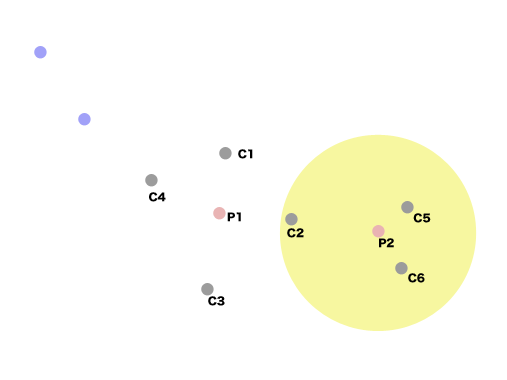
同様に行う(図の左側)。
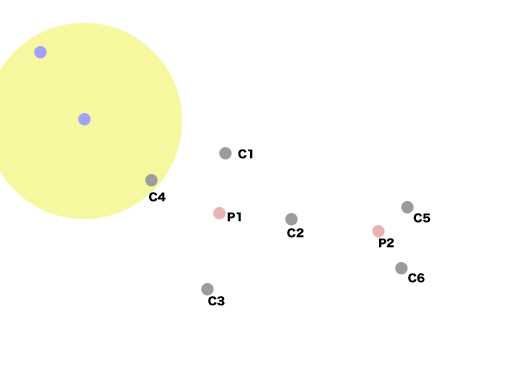
これもC4 -> P3とつなげることができる。
最終的に全部つなげることができた。
実際に階層型の木を描くと以下のようになる。
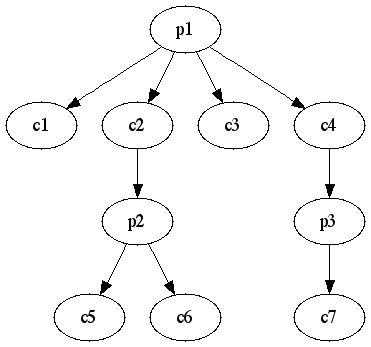
単純だけど、全体の構造を保ったままCytoscapeで眺められていいかなーと思うんだけど。
もっといい方法あったら教えてもらえると嬉しいです。
