06082010 chemoinformatics Python cytoscape
chemoinformaticsでもネットワークアナリシスは重要だと思う。今はマイナーだけど今後精力的に研究されんじゃないかなぁと。
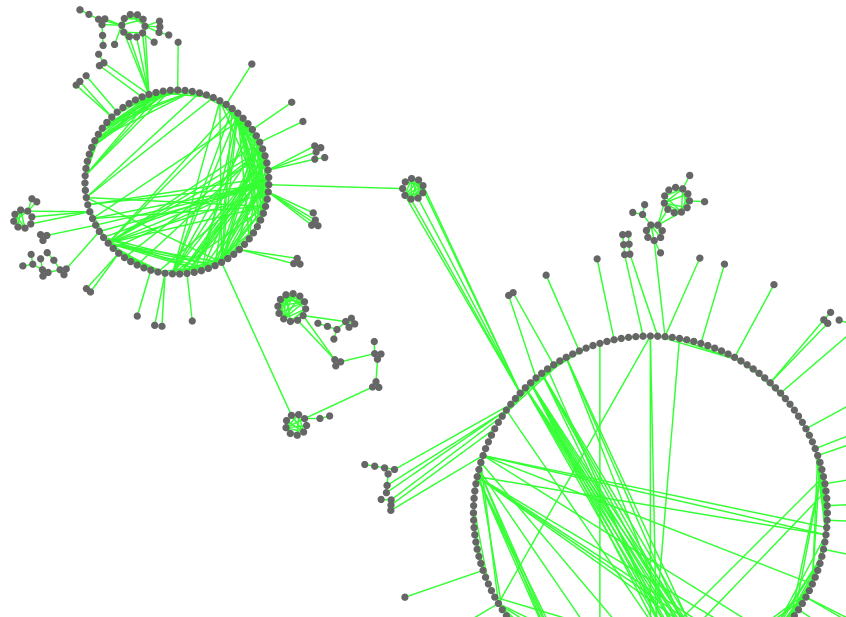
pybel使うとシミラリティベースのネットワークは簡単に作れる。
import pybel
mols = list(pybel.readfile("sdf", "pc_sample.sdf"))
fps = [x.calcfp() for x in mols]
for i in range(len(fps)):
ids = []
for j in range(i,len(fps)):
if (fps[i] | fps[j]) > 0.5 and i != j:
ids.append(mols[j].title)
print "%s %s %s" % (mols[i].title, "sim", " ".join(ids))
SIF(simple interaction format)形式なので、拡張子sifでcytoscapeに読み込まさせればOK。
あとでケモインフォクックブックにも載せておこう。