08032020 chemoinformatics bioinformatics
COVID-19で色々と大変な状況になったので、開催を中止することにしました。
かなC
日程決まりました。3/28(sat)です。
参加登録は以下からどうぞ
演題も募集中です。
08032020 chemoinformatics bioinformatics
かなC
日程決まりました。3/28(sat)です。
参加登録は以下からどうぞ
演題も募集中です。
10012020 chemoinformatics
25を入れたときのログ。今回は高速化のために postgres.confのfsyncをoff にしておいた。
dropdb chembl_23 psql -l createdb chembl_25 psql -l pg_ctl -D /usr/local/var/postgres stop # vim postgres.conf -> fsync=off pg_ctl -D /usr/local/var/postgres -l logfile start $ pg_restore -j 2 -d chembl_25_postgresql.dmp
28122019 chemoinformatics
This is the 24th article of the Drug Discovery Advent Calendar (Dry) 2019.
I was very impressed by the weak hydrogen bonds in the article, A C-H···O Interaction Against IRAK4 / Drug Hunter . So I checked that interaction with Fragment Orbital Method
PDB:6UYA was used as a complex structure and details of calculation are here
If you are not familiar with FMO, this book will help you.
 Fragment Molecular Obital Method : Practical Applications To Large Molecular Systems [Hardcover] [Jan 01, 2009] Fedorov
Fragment Molecular Obital Method : Practical Applications To Large Molecular Systems [Hardcover] [Jan 01, 2009] FedorovThis is actually the article for the 24th advent calendar, but I can't give you this book because I'm not Santa Claus. If you click on the link instead, I'm sure that Amazon will deliver this book to you instead of Santa Claus.
What a wonderful Christmas gift it is! Now, click it!
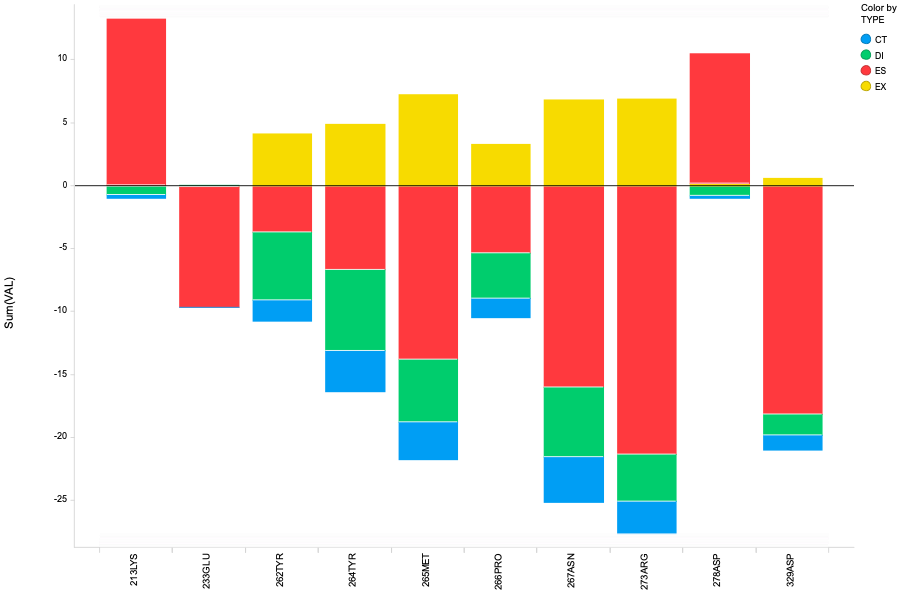
The result is here. I only show the interaction which have strong stable/unstable interactions for clarify.
The result shows that there is a C-H···O Interaction between compound and protein(TYR264 in this case), but the interaction energy is rather weaker than the typical hydrogen bond.
*** The amino acid number and fragment number doesn't exactly match in FMO. The carboxylic acid moves to the next residue because of cleaverage in protein at C (sp3) -C (sp3) bond.
And also I'm not sure that the nature of this interaction is same as hydrogen bond. I guess it also includes side chain effects because DI term is slightly large. To clarify this, we can replace TYR with Gly and execute FMO calculation again.
Next, we will interpret it by frontier orbital, but it's WIP.
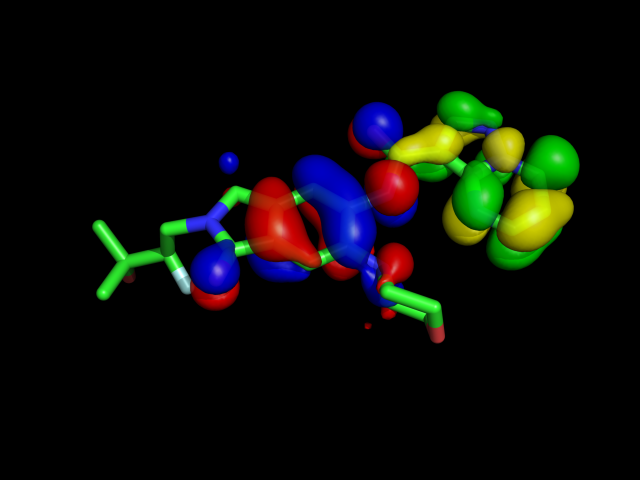
I guess we'll know a lot if I do it so far, but I've run out.
Have a happy new year!
15122019 chemoinformatics
この記事は、創薬 (dry) Advent Calendar 2019 の14日目の記事です。
先日とある学会で@torusengokuのDNAメチル化のポスターを聞いていたら、横からある助教の人が乱入してきて「メチル基とアミノ酸残基の相互作用がこんなに強いのは納得いかない」といようなことを言い出してディスカッションになった気がします。結局これはカチオン-πであろうということで終結したのだけど、この議論を通して、
という重要な知見を得ることができました。ついでに@torusengokuからMeCP2も同じような結合様式を取っているようだと教えてもらったので、計算してみたというのがこのエントリーになります。
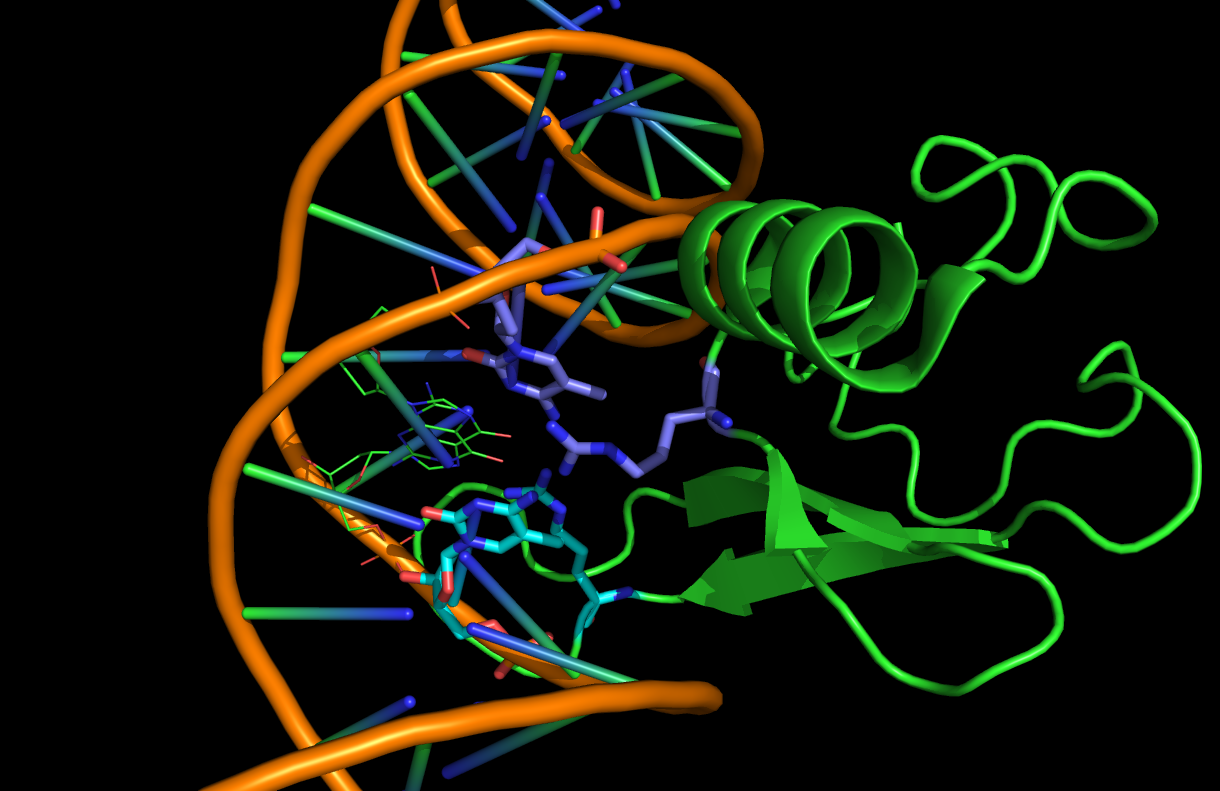
MeCP2はmethyl-CpG binding protein 2であり、methyl-CpGを特異的に認識して結合するタンパク質です。結晶構造からはMeCPのARGがメチル化されたCの対のGと結合しています。この相互作用は相補鎖についても同様です。
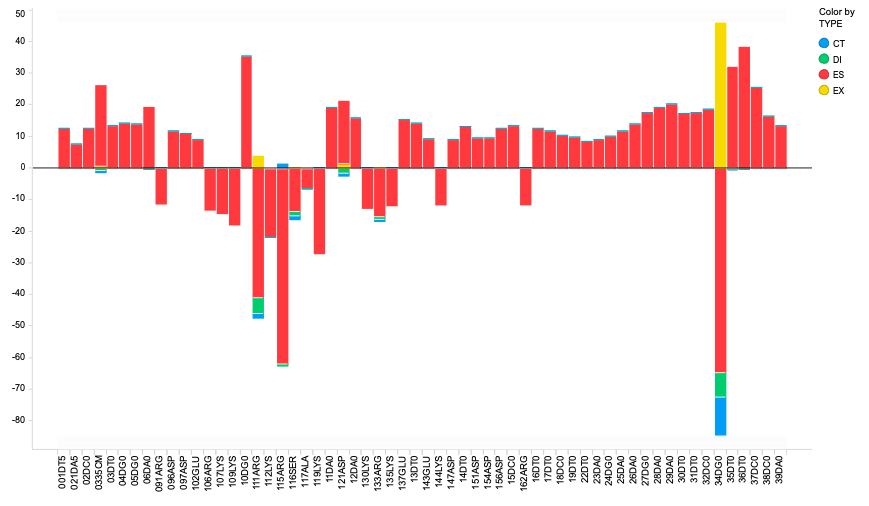
FMO計算をしてメチル化CのPIEDAを見てみると、ES項がごちゃごちゃと現れてきますが、実際の溶液中だとほとんどキャンセリングしてると考えられます。このあたり改善していく必要があると思いますが、どうすればいいのかわからない。
ESを除いて見やすくすると、対となっているGとの相互作用が強く出ていますが、それと同時にARGとのDIの相互作用もかなり強く出ています。これはカチオン-π相互作用でしょう。
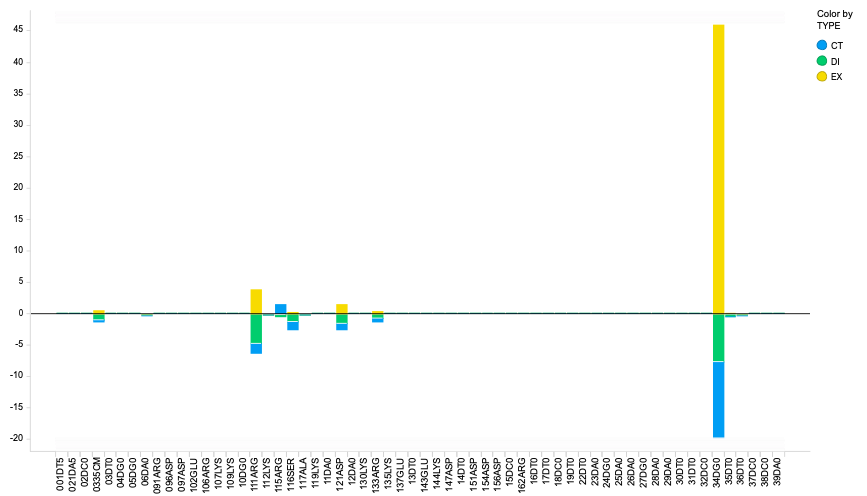
このあとは脱メチル化したモデルを作ってPIEDAの差分を取ったりするとメチル化の効果によりどのくらい結合が安定化したかとか成分がどう変化したかとかの詳細がわかります。
このような、protein-DNA interactionやprotein-protein interactionに関する理解を深めておけば、従来のkey-lock型の相互作用に基づかない新たな生体干渉方法のアイデアが浮かぶんじゃないかなーと思います。
先週の分生の相分離のワークショップ出たときも、「相分離はエンタルピードリブンだ!」とおっしゃっていたので、構造生物学から攻めることできちんと理解される生命現象は少なくないのではないかと考えています。
14122019 chemoinformatics
なにげに構造活性相関シンポジウムは初めてです。依頼講演で呼ばれたついでにポスター発表しようと思ったらオーラルに変更されたので、2演題話してきました。両方とも活発な質疑ができたので、少しは盛り上げられたかな?と思いました。
構造活性相関シンポジウムって創農薬寄りなんですね。藤田センセだからまぁそうなんだろうなとは思いましたが。全体的に参加者の人達はMachine Learningをもうちょっとしっかり理解しておいたほうがよいのではないかな?と感じました。個人的にはMol2Vecの面白い使い方を知れてよかったのと私のにわか量子化学力をシャキッとさせるコネクションができて満足した学会でした。
あと、ポスターにOCamlのロゴを印字している方がいてOcaml愛に溢れとるなーと感じた。
私もHaskellでコード書いてポスターにロゴをのっけたいところ
前泊入りしたので羽田空港にて。静岡のHaskellerの嗜み。

熊本についたら、なんかイベントやってた。

夜はライフイズジャーニーっていうお店で熊本ラーメン食べた。

つまむものがないので、ラーメンをつまみに焼酎飲むらしい。まわりがそんな感じだったので 私もそれに倣った


帰りにくまモンラベルの白岳を買った

学会会場は熊本城の近くだったので、ちょっと早めに到着して散策(ポケストップを回す)してみた。


懇親会では日本酒飲めたので満足。あと色々お話したり議論できてよかったです。トレーナーのフレンドも増えたw

次の日も発表なので二次会に出ずにホテルに戻ったけど、お腹が空いたのでラーメン注入。 ここはまぁまぁかな。


二日目のランチは、前職で一緒に仕事をしていた方とラーメン。天外天行ったら夜のみ営業だったので黒亭。 生卵は入れず。


学会終了しても終電に間に合わないのでもう一泊して馬った。


馬ホルモン焼きとホルモンのポン酢和え。


馬刺しは別に興味なかったのでこんな感じ。御殿場でいつでも買えるしね。

最後にまたライフイズジャーニーに寄った。美味しいね、ここ。

今回は博多ラーメンを注文。こっちのほうが好きかな。


08122019 chemoinformatics
この記事は、創薬 (dry) Advent Calendar 2019 の8日目の記事です。
皆さん料理は好きですか?レシピをいくつ覚えていますか? 食材や調味料が足りなかった場合にあなたはそれらレシピをアレンジすることができますか? もしアレンジできないのであれば、そもそも料理というものを理解していない証拠なので「料理の四面体」を読みましょう。
読んでいない人のためにネタバレを用意したので、ざっと読んでみてください。ちなみに私の強烈なaha体験はアヒージョと茹でた肉や野菜の油がけは可換であるという事実でした。これによりメタレシピというものに目覚めたし、急激に料理の応用の幅が広がった気がします。
さて、ここからが本番です。
みなさんはモダリティをご存知でしょうか? もし知らなければNew Modalities, Technologies, and Partnerships in Probe and Lead Generationが参考になると思います。
以下は全文にアクセスできますが、論調が私の主張と合わない部分があるので参考として載せておきます。
モダリティの話が出てくると文脈には「低分子はオワコン、これからは中分子の時代」という既存の手法との対立が含まれていたり、百貨店よろしく、いくつのモダリティをピラーにするかという風に語られることが多いように思いますがもう少し統一的な視点を与えられないかというのが今回のお話です。
そこで冒頭の料理の四面体の枠組みを借りてきて、それぞれの辺に「分子量」「MoA」「測定系」というパラメータをアサインします。するとそれぞれの辺を頂点に持つ三角形が(広義の)モダリティを形成することになります。
分子量に関してはよいし、MoAもセントラル・ドグマだと考えればOKかなと思いますが、測定系が連続値をとるというのはちょっと乱暴だけど無視してください。
これでキレイに抽象化できたかなと。
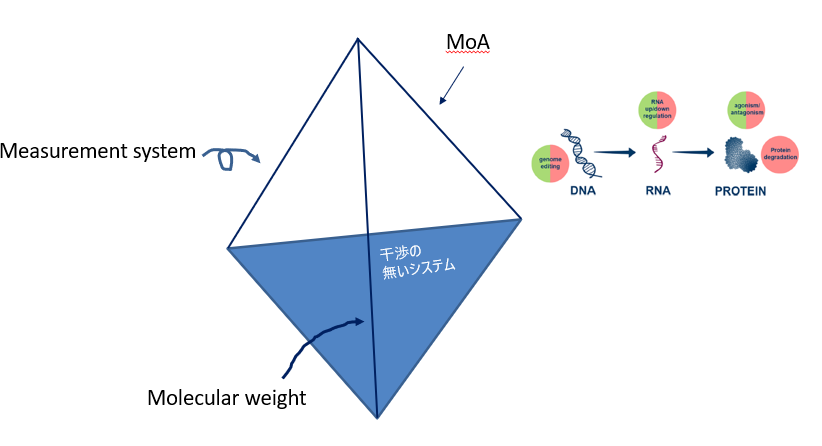
こうしてみる特にモダリティで組織化する場合にはそれぞれのモダリティに応じたテーマの探索を志向すると思うのでプロダクトアウト色が強くなりがちだと思うのですが、そのあたりどういう風に舵を取るんでしょうね? 個人的にはRight Target -> Right Modalityをどう選ぶかというMoA in(マーケットイン)なアプローチは重要なんじゃないかなーと思います。そしてそれができるのがDryの人達ではないかなと考えています。
結局モダリティで組織化せざるを得ないのは技術ロックされるからだけど、コンピューターサイエンスとしては低分子の構造最適化も、ペプチドの最適化に特に壁はないし、バイオインフォマティクスもやればいいので、別にプロダクトアウトの立場になる必要性もないでしょう。むしろRight Targetが得られたあとに適切なモダリティの選択とドラッグデザインができれば楽しくプロジェクトに関われてハッピーなんじゃないかなと。
ただそのためには、key-lockシステムを超えたMoA干渉のアプローチ方法を理解しておく必要があるかなぁと思います。さらには新しいWetの技術などもキャッチアップしないといけないけど、分子設計者としての幅は広がって楽しいでしょう。
今回昔書いた以下のエントリを参考に書き直してみました。
07122019 chemoinformatics bioinformatics
朝は粥

分子生物学会というなのお食事会も最終日

昼は海鳴でラーメン。たしかにここは美味しかった。博多滞在中結局2杯しか食べられなかったので 今度はラーメン詣でにいこうかな。

デイトスの酒屋の角打ちでスタバったら当たりをひいて一杯サービスになった。


このまま帰るとただ酒になってしまうので、みやざきのひでじビールを頂いた。

新幹線の中では先程の酒屋で購入した三井の寿をちまちまやった。新幹線で飲まないと立派なHaskellerにはなれない。 すくなくとも静岡では一人前のHaskellerとしては認められない。

05122019 chemoinformatics bioinformatics 博多
05122019 chemoinformatics bioinformatics 博多