15 12 2019 chemoinformatics Tweet
この記事は、創薬 (dry) Advent Calendar 2019 の14日目の記事です。
先日とある学会で@torusengokuのDNAメチル化のポスターを聞いていたら、横からある助教の人が乱入してきて「メチル基とアミノ酸残基の相互作用がこんなに強いのは納得いかない」といようなことを言い出してディスカッションになった気がします。結局これはカチオン-πであろうということで終結したのだけど、この議論を通して、
- DNAとタンパク質の相互作用って、プリン体という低分子とアミノ酸の相互作用に還元できるんだ
- エピジェネってエントロピードリブンだと思って近づかなかったけど、実はエンタルピードリブンで構造生物学向きなんだ
という重要な知見を得ることができました。ついでに@torusengokuからMeCP2も同じような結合様式を取っているようだと教えてもらったので、計算してみたというのがこのエントリーになります。
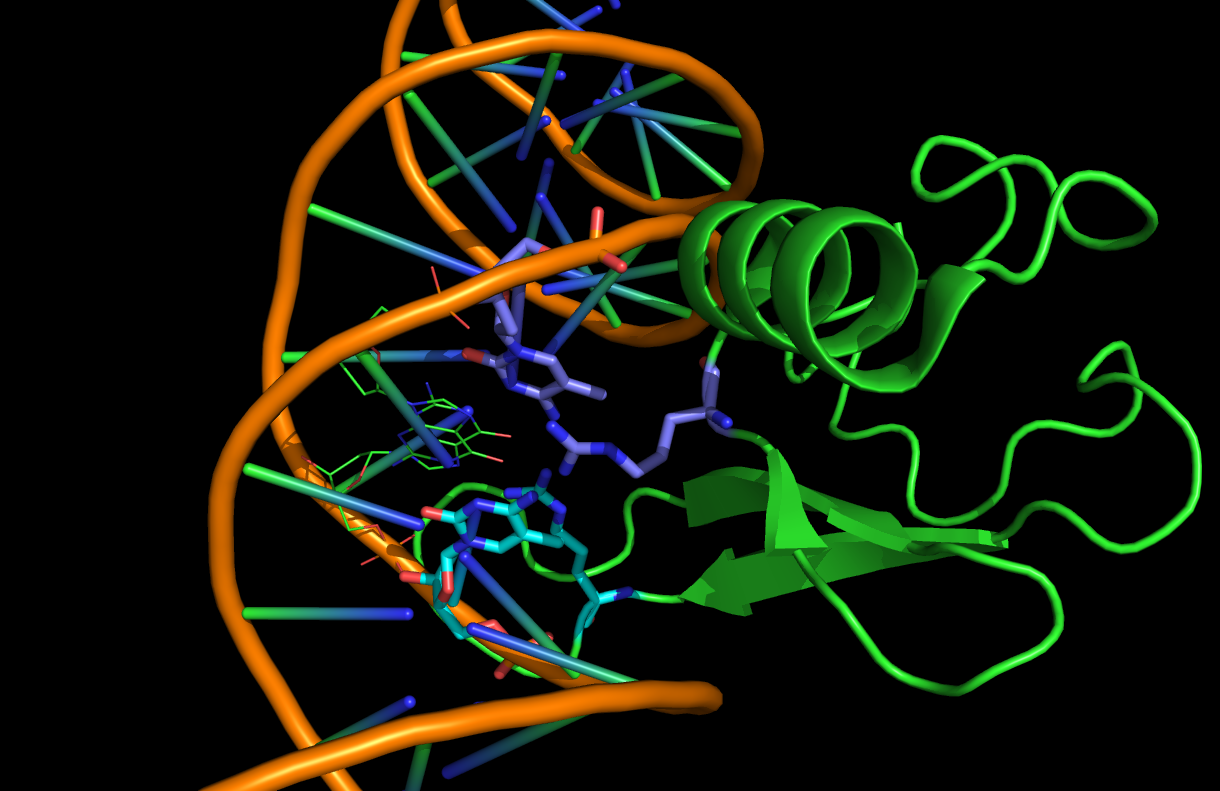
MeCP2はmethyl-CpG binding protein 2であり、methyl-CpGを特異的に認識して結合するタンパク質です。結晶構造からはMeCPのARGがメチル化されたCの対のGと結合しています。この相互作用は相補鎖についても同様です。
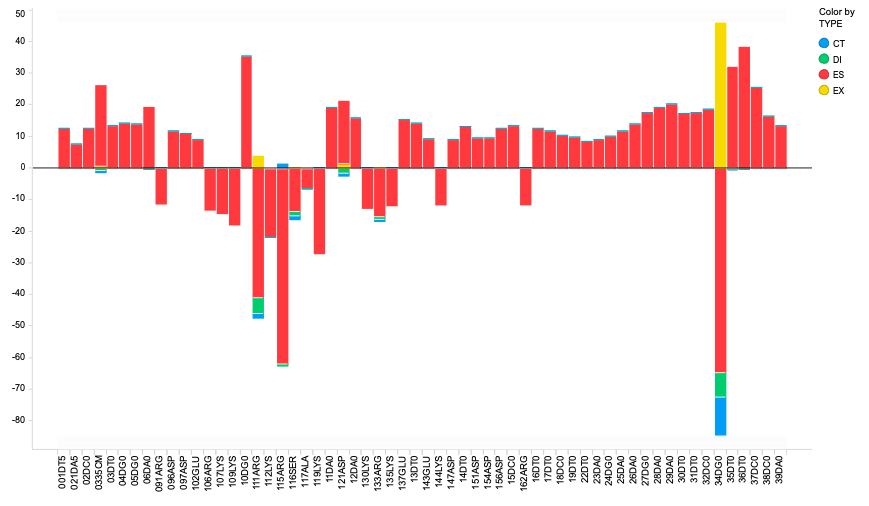
FMO計算をしてメチル化CのPIEDAを見てみると、ES項がごちゃごちゃと現れてきますが、実際の溶液中だとほとんどキャンセリングしてると考えられます。このあたり改善していく必要があると思いますが、どうすればいいのかわからない。
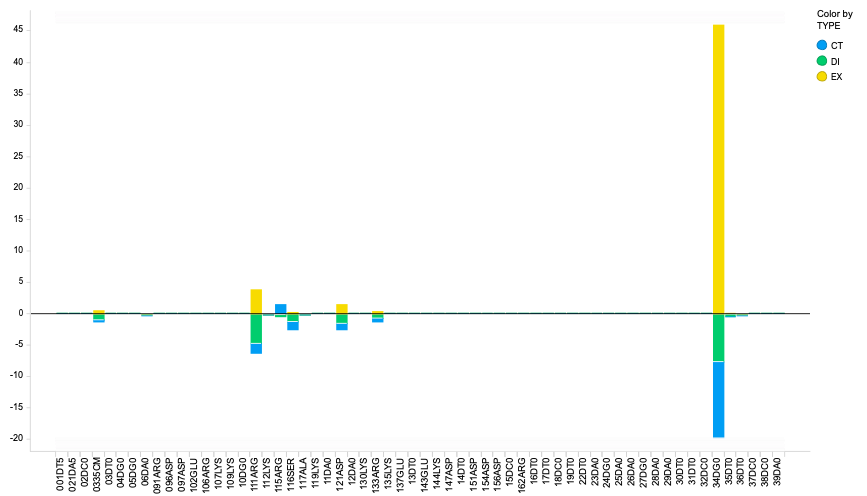
ESを除いて見やすくすると、対となっているGとの相互作用が強く出ていますが、それと同時にARGとのDIの相互作用もかなり強く出ています。これはカチオン-π相互作用でしょう。
このあとは脱メチル化したモデルを作ってPIEDAの差分を取ったりするとメチル化の効果によりどのくらい結合が安定化したかとか成分がどう変化したかとかの詳細がわかります。
このような、protein-DNA interactionやprotein-protein interactionに関する理解を深めておけば、従来のkey-lock型の相互作用に基づかない新たな生体干渉方法のアイデアが浮かぶんじゃないかなーと思います。
先週の分生の相分離のワークショップ出たときも、「相分離はエンタルピードリブンだ!」とおっしゃっていたので、構造生物学から攻めることできちんと理解される生命現象は少なくないのではないかと考えています。
