プロジェクトの成功というのは個人のスキルの継続的な向上に加えて、チームのコミュニケーションを如何に円滑にするかにかかっているかと思う。
まぁ、それは創薬プロジェクトでも一緒なわけで。でコミュニケーションを円滑にするためにそれ用のツールに頼るのがいいかと。
- レポート、ガントチャートが自動で作成される
- 適切なワークフロー管理
- トレーサビリティの向上
- 他のツールとの連携(例えば化合物管理システムとかELNとか)
なんかができるのがいいかなと思っている。特に化合物管理システムについては5Wの最後のWHYを記録できないのが問題で、これを記録するような簡単な仕組みがないとコミュニケーションの円滑化につながらないような気がする。
- WHO: OK
- WHEN:OK
- WHAT:OK
- WHERE,HOW: OK(ELN)
- WHY:NO
週に一度会議をするとかは一見良い案に見えるけど、現状把握だけで時間を取られるし、そのためのレポート作成時間もバカにならないし、結構コスト高なのではないかなと。
こういうあたりはredmineのようなITSで管理すれば、割りとうまく回りやすいのかなと思っている。というより一人で使っているけど、調子いい。
脱Excel! Redmineでアジャイル開発を楽々管理のフローは創薬のサイクルでも大体同じ流れですね。
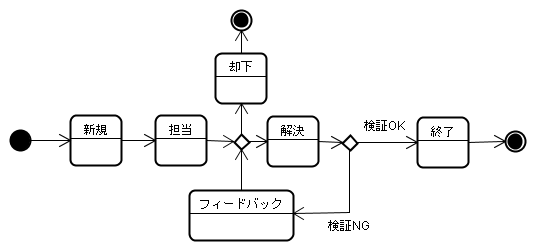
- 合成案をだして
- 担当者決めて合成
- アッセイ結果をフィードバック
まぁPDCAサイクルまわすようなのは大体こんな感じかと。
バックログとしての合成案
AZは基本的にはこの考え方だと思います。
アジャイルな創薬ということを考えたらフィードバック結果で合成計画を柔軟に変更できないといけないから、バックログを持っていたほうがいいんだろうなと思う。今の日本だと、合成案考えたヒトが合成するみたいな風潮になっていて、間にレビューもおかないし、柔軟性が足りないし、ケミストの能力がそのままストレートに反映されるのでそれっていいのかなーと思ったりもする。あとは、合成案の批判=ケミストの人格批判みたいに受け取るヒトが多いというのも、案は案として客観的に評価できてない証拠だよねーと。
というわけで、こういったあたりに興味があれば参加してくださると嬉しいです。
(2013.09.27 追記) Redmineの操作に関してはプログラミングのスキルは必要ありませんのでお気軽に(Q&Aに追記しておきました)。
ちなみに懇親会は三島バルという飲み歩きイベントに便乗しまして、二次会どこかに集合してまったりやりたいなと考えています。
参考書籍
 チケット駆動開発
チケット駆動開発
小川 明彦
翔泳社 / 3444円 ( 2012-08-24 )

 メディシナルケミストリー
メディシナルケミストリー







 アジャイル開発とスクラム 顧客・技術・経営をつなぐ協調的ソフトウェア開発マネジメント
アジャイル開発とスクラム 顧客・技術・経営をつなぐ協調的ソフトウェア開発マネジメント チケット駆動開発
チケット駆動開発

