09 07 2013 chemoinformatics Python Tweet
pychembldb使えば楽勝だというということの証明をしようと思ったが、意外に面倒くさかった。
- ヒトのアッセイ系
- 信頼レベルマックス(Direct single protein target assigned)
- アッセイのタイプはBinding
という条件でデータを引っ張ってくる。その後構造数<2のファイル(MMPにならない)を削除して、メタデータ(アッセイID, Uniprotのアクセッション番号、一般名称、データ元のジャーナル)を吐き出したあと、活性データをTSVに出力するようにしている。
最初はsdfのほうに活性情報も付けておけば楽勝じゃないかと思ったが、スキーマ見てたら測定タイプが正規化されてないうえに、AssayじゃなくてActivityのほうについてることに嫌な予感がしたので調べた。
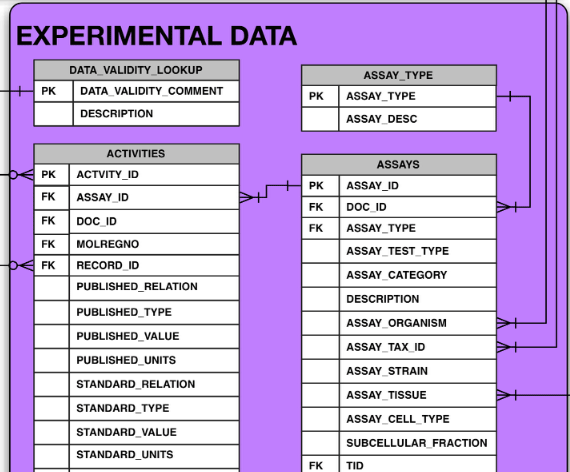
やはり、pIC50とIC50が混在してたり、InhibitionとIC50が混在していた。
これはペアに出来ないので僕の場合はpandasでゴニョるのでこうしましたが、PPのスキルが高まっていてこっちでやれるのであればsdfに活性入れておいたほうが取り回しやすいかも。
コードはexamples/recreation.pyにあります。ファイル名があれなのは今朝コードを買いている時にスーパーカーを聴きまくっていたからというわけなので察してください。
