26 06 2013 chemoinformatics Tweet
VAMMPIREという
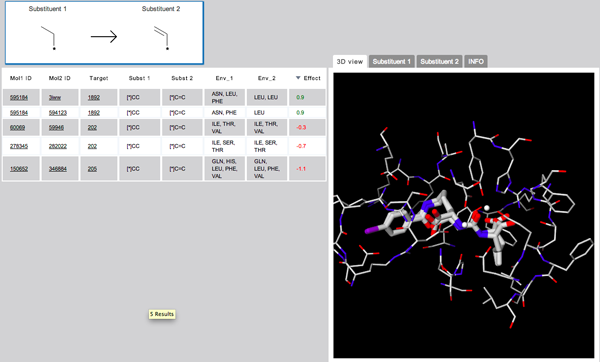
- PDBの複合体構造からリガンドを引っ張ってきて
- ChEMBLからMMPをサーチして
- ドッキングモデルを構築してWebGLで表示する
っていうDatabaseの論文をみつけたのだけど、コンテキストが入りまくりなのはどうなのかなぁ?タンパク質-リガンド相互作用を骨格の多様性で積分したら普通ゼロになるよね?実用性に疑問符がと思ったが、
ふと、あーFMOの題材としてはいいんじゃないのかなぁと閃いた。置換基変換のペアとKi差や活性差の値が出ているからFMOのエネルギー比較して色々遊べるかな。