03072008 chemoinformatics R
一通り読んだ。分かりやすくてお薦めの一冊。
まぁ、なんというか僕はクラスタリングとかPLS,PCRなんかのほうに偏っていて検定とか苦手(というよりもあまり使わない)のでこういったタイプの本は勉強するによいし、手元においておくと何かと便利(なはず)
03072008 chemoinformatics R
一通り読んだ。分かりやすくてお薦めの一冊。
まぁ、なんというか僕はクラスタリングとかPLS,PCRなんかのほうに偏っていて検定とか苦手(というよりもあまり使わない)のでこういったタイプの本は勉強するによいし、手元においておくと何かと便利(なはず)
30062008 chemoinformatics jython opsin
jython+web.pyがお手軽で、ちょっとしたことをやるならいい感じ。
jythonのosモジュールにはfstatがないのでtrunkのSimpleHTTPServerの静的ファイルの転送ができない。そのため、2.2.1のSimpleHTTPServerと入れ替えた。
import java.io.StringReader as StringReader
import org.openscience.cdk.interfaces.IMolecule
import org.openscience.cdk.io.CMLReader as CMLReader
import org.openscience.cdk.ChemFile as ChemFile
import org.openscience.cdk.layout.StructureDiagramGenerator as StructureDiagramGenerator
import uk.ac.cam.ch.wwmm.opsin as opsin
import net.sf.structure.cdk.util.ImageKit as ImageKit
import web
urls = (
'/(.*)', 'img2d'
)
class img2d:
def GET(self, name):
cml = opsin.NameToStructure().parseToCML(name).toXML()
str_reader = StringReader(cml);
cmlr = CMLReader()
cmlr.setReader(str_reader)
chem = cmlr.read(ChemFile());
mol = chem.getChemSequence(0).getChemModel(0).getSetOfMolecules().getMolecule(0)
sdg = StructureDiagramGenerator()
sdg.setMolecule(mol)
sdg.generateCoordinates()
mol = sdg.getMolecule()
ImageKit.writePNG(mol, 300, 300, "./static/test.png")
print '<h1>' + name + '</h1>' + '<img src="/static/test.png" />'
if __name__ == "__main__": web.run(urls, globals())
http://localhost:8080/(2,3-diethyl-benzyl)-isobutanolというURLにアクセスすると、IUPACを2次元構造に変換していい感じに描画して表示してくれる。
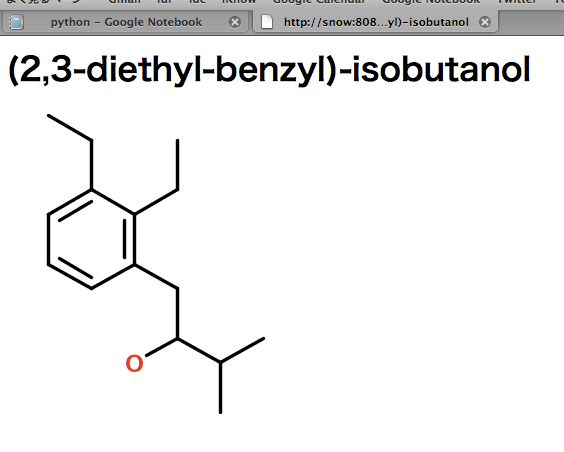
ImageKitが必ずファイルに出力するのでテンポラリのファイルを作ればいいのだけど、とりあえず動く事を確認したかったので決めうちの名前で。
29062008 chemoinformatics life
かなり消化不良だが読み終えた。
一応、僕はデザイン関係ではなくて、製薬の企業でデータマイニングとかの研究がメイン。マイニングするためのデータが欲しいという理由で、webのシステム組んだり、データを集約するための仕組みをあれこれ考えたりとかしている(今は)。だから、ターゲットとしている読者からは離れているかもしれない。
概要は正直辛かった。バックグラウンドが乏しいので、「誰の何とかが提唱したなんとかは」みたいに書かれても、なかなか自分の中でつながっていく感がなかった。ある程度分かっているヒトが読むとつながっていく感が感じられて面白いんだろうなとは思ったんだけど。第一部の概要は読んでて辛かったので途中で投げ出した、二週間くらい。他の本(ロベールとか)読んでて読むものがなくなってまた読み始めたって感じだった。
で、実践編はなかなか面白かった。ポストイットを張りながら読んだ。こっちを読んで考えながら一部の概論に立ち戻るという読み方が正解だったかなと、今は思う。
結局ペルソナっていうのは、TDDでのテストとかBDDの仕様に近いもんなのかなと。安心して戻ってこられるよりどころみたいな。
昔考えた人工無脳ケミストもそういった観点で捉え直せば、ケモインフォマティクスのためのアジャイルプロセス用ツールとして使えるかもしれんなと思った。
参考文献の中でおもしろそうなのを読んでいこうかな。
29062008 chemoinformatics jython opsin
CherryPyはSignalがないぜよっていうエラーを解決できなさそうだったのであきらめた。
webpyはSSL関係のクラスをコメントアウトして830行あたりをちょっと修正すれば動く。
これで、cgiとかサーブレットみたいな面倒な手段をとらなくても、javaライブラリとwebserverをつなげられる。
例としてIUPAC名をCMLに変換するjavaライブラリであるopsinを利用したwebappを作る
import uk.ac.cam.ch.wwmm.opsin as opsin
import web
urls = (
'/(.*)', 'hello'
)
class hello:
def GET(self, name):
i = web.input(times=1)
for c in range(int(i.times)):
print opsin.NameToStructure().parseToCML(name).toXML()
if __name__ == "__main__": web.run(urls, globals())
http://localhost:8080/benzeneってアクセスするとbenzeneのCMLがブラウザに出力される。
17062008 chemoinformatics
13042008 chemoinformatics bioinformatics wii
ずいぶん前に加速度センサでPyMolを動かしてみたことがあるんだけど、案の定、
細かい制御が出来ないから使えねーよな、プレゼンだったら別にいいんだけどよ
みたいなコメントが入っていて、やっぱワイヤレスなセンサーバーが欲しいなあと買ってしまった。
これで、PyMolいじりだ。今回使用したMoleculeはMolecule of the MonthにのっていたAdrenergic Receptorsだ。ちなみに、僕はTopoisomerasesとかが気に入っているが。
IRセンサーのほうが位置を決めやすいので動かすのがやっぱ楽チン。
IRセンサーと加速度センサーの入力を取りたいだけだったらbluetoothでやりとりできるWiiコントローラのほうがGainerよりも楽だな。
08042008 chemoinformatics bioinformatics
PDBWikiというのが立ち上がったらしい。どういうアノテーションが溜まっていくのか興味あり。
とか書いてたら、MDとかMOとかのアノテーションがついた結晶構造のデータベースがあってもいいじゃないかと思った。PDBのデータを見ていて思うのはインタラクションに関するデータがないのがアレかなと思ってたりするので。リガンドとたんぱく質の相互作用に関する知識を深めていけるような何かがあればいいなぁ。
PDB-631Gプロジェクトとかそんなかんじの。
またはMolecule of the Month みたいな感じで、Interaction of the Monthとか。
22032008 chemoinformatics
ふむ。でもあんまり便利感がないなー。っだって、長い番号打ち込まないといけないんでしょ?
構造式のgooglesuggestみたいなのがあればいいんだけどね。
18032008 chemoinformatics perl bioinformatics
文献はpdfを自作のイントラソーシャルブックマークサービスっぽいのを使って管理しているのだけど、pubmedにのるよりもペーパーの方が早かったりすることのほうが多く、pmidをつけることが出来なくてもどかしい思いをしてばかりいる。
use strict;
use warnings;
use CAM::PDF;
my $pdfname = $ARGV[0];
my $pdf = CAM::PDF->new($pdfname);
my $page1 = $pdf->getPageContent(1);
my ($result) = $page1 =~ m!\d+\.\d+\/.+?(?=\))!gx;
print $result if $result;
抽出したdoiをeutilsに問い合わせて、あとは適度な塩加減を加えつつゴニョルとよさげ。
で、そのソーシャルブックマークサービスはpmidをプライマリキーにしてるもんだから始末に負えない、というかそのうち、匠的にリフォームする気概で、今日もだましだまし使った。
09012008 chemoinformatics
SBDD(Structure-Based Drug Design)って、結局モデルの精度にあわせてMDとかMOとか視点を変えなきゃいけないからマルチパラダイムだよなと思った。
あとそろそろ単なるMO計算も飽きてきたので、今年はMO計算とQSARをうまいこと組み合わせた手法とか編み出してみたい。あと、ケミストの合成ロジックをエミュレートするような無限ストリーム(仮)を実装したい。
あーでも、他のことに興味がいっちゃうんだよなー。