31032007 bioinformatics turbogears
ふと、毒性評価のチームが「病理とかの画像にタグつけて管理したいなぁ」とのたまうので、
ほう、それはイントラflickrクローンみたいなものに落書き機能をつけてタグとコメントで管理できる洒落たツールが欲しいということか?
それいいじゃん!ということでいけそうか試してみた。
Making a Flickr Killer With TurboGearsを参考に喜久酔ひっかけながらもそもそと手を動かすこと30分。
おーCRUDくらいまでできた。
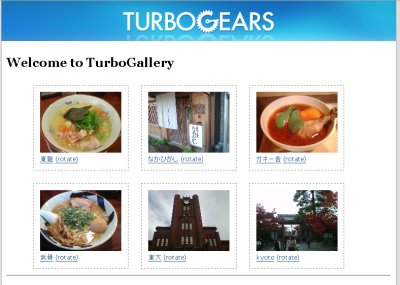
SQLiteのText型ってBLOB突っ込めんの知らんかったが、実際はバイナリはデータベースに突っ込むよりはファイル管理のほうがいいのかな。タグ付けは自分で書いてもいいし、Tastyにお任せしてもいい気がする。
落書き機能(病理画像に着目ポイントを書き込む機能)はAjaxかAS3でつくる。
- http://nanto.asablo.jp/blog/2005/09/27/89628
- http://josephlabrecque.com/archives/17
- http://www.jamesward.org/wordpress/?p=66
gimpとかphotoshopのレイヤーみたいに重ねる感じにすればいいと思うんだけど。そのうち、まとまった時間をとってやる。

 ライフサイクル イノベーション 成熟市場+コモディティ化に効く 14のイノベーション
ライフサイクル イノベーション 成熟市場+コモディティ化に効く 14のイノベーション バイオプログラミング―バイオインフォマティクス演習 (Ohm bio science books)
バイオプログラミング―バイオインフォマティクス演習 (Ohm bio science books)