普段は仮想の空間(ケミカルスペース)とか多変量の解析ばかりしていて、リアルな地理情報を扱うようなエリアマーケティング的なところにはタッチしたことはなかったんだが、フジブログにインスパイアされてGISにも手を出し始めた。
やっぱ身近な話題は面白いね。データは政府統計のH12年国勢調査(小地域)をつかった。
library(maptools) library(RColorBrewer) library(classInt) f <- readShapePoly("h12ka22210.shp") pop <- f$JINKO / f$AREA * 1000 colors <- brewer.pal(9, "YlOrRd") brks<-classIntervals(pop, n=10, style="quantile") brks<- brks$brks plot(f, col=colors[findInterval(pop, brks,all.inside=TRUE)], axes=F)
密度はf$JINKO / f$AREA * 1000で計算しているので単位面積あたりの人口ということになりますね。フジブログでは単純に人数をカウントしてるだけですが、実際には地域の何%が流出入したかをかんがえるべきかなと思います(予算配分的には人数でカウントするのが好ましいのかもしれないが)。
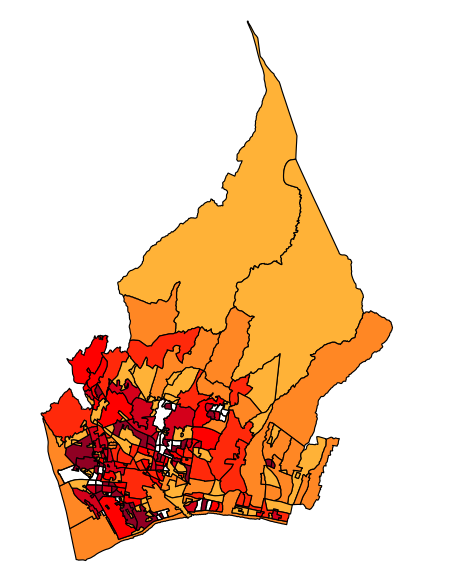
既にデータは国が出してんだからこういうのを小学校とか中学校のカリキュラムに取り入れれば、算数、地理、歴史の教育になっていいと思うんだけどなぁ。Rもオープンソースだしね。

 グラフィックスのためのRプログラミング
グラフィックスのためのRプログラミング グラフィックスのためのRプログラミング
グラフィックスのためのRプログラミング The Grammar of Graphics (Statistics and Computing)
The Grammar of Graphics (Statistics and Computing) Rクックブック
Rクックブック R言語逆引きハンドブック
R言語逆引きハンドブック グラフィックスのためのRプログラミング―ggplot2入門
グラフィックスのためのRプログラミング―ggplot2入門 デジタル画像処理 (Rで学ぶデータサイエンス 11)
デジタル画像処理 (Rで学ぶデータサイエンス 11)

 Rパッケージガイドブック
Rパッケージガイドブック Perl CPANモジュールガイド
Perl CPANモジュールガイド R Cookbook
R Cookbook Rによるバイオインフォマティクスデータ解析 第2版 -Bioconductorを用いたゲノムスケールのデータマイニング-
Rによるバイオインフォマティクスデータ解析 第2版 -Bioconductorを用いたゲノムスケールのデータマイニング-