沼津ナイトマーケット2016に行ってきた。
ベアードビールの沼津ラガーを飲みながら狩野川沿いで涼んでた。気持ちいい


座ってる場所の直ぐ側にポケストップがあったので当然ルアー挿してポケモン狩りもビールと並行して実施
カメックスが飛び出してきて、息子狂喜乱舞。その後モンスターボールが枯渇して発狂、帰るまでいじけてたw

日頃から地道にポケストップは回しておかないとダメだという教訓ですな。
沼津ナイトマーケット2016に行ってきた。
ベアードビールの沼津ラガーを飲みながら狩野川沿いで涼んでた。気持ちいい


座ってる場所の直ぐ側にポケストップがあったので当然ルアー挿してポケモン狩りもビールと並行して実施
カメックスが飛び出してきて、息子狂喜乱舞。その後モンスターボールが枯渇して発狂、帰るまでいじけてたw

日頃から地道にポケストップは回しておかないとダメだという教訓ですな。
07082016 pokemongo
Lv22の経験値が7割くらいたまったところで、(ちょっとめんどくさくなって)しあわせタマゴブースト使って色々進化させた。ヤドランとアーボックは図鑑を埋められたので良し。
ジム戦でナッシーに何をぶつけたらいいのか悩む。炎タイプはシャワーズでゴリ押しできるので楽なんだけどその後にブースター控えていると結構困る。ブースターは相性的にいいはずなんだけど意外にサクッと倒されちゃうし、現状はより強い手持ちのナッシーぶつけているけど、虫タイプや、ほのおタイプの低CPでなにかいないのかなぁ。
このレベルになってくると家の周りをウロウロしても図鑑がほとんど埋まらない。埋めるためには違う場所に積極的に遠征しないといけないが、Ingressほど新規ポケストップ(ポータル)をまわすモチベーションがない。レベル上げだけだと経験値を稼ぐだけの作業ゲームになってしまうのでこれもまたモチベーションが保てない(Ingress lv14で力尽きたしw)。というわけで、レベルを重視するか図鑑のコンプを目指していくのか悩ましいところだ。
どうしようかなぁ。
息子のラーメンに浮かぶ卵を守るポッポ。鮎沢Pにて

07082016 沼津 snorkeling
今年はなんか忙しくて週末が潰れまくっているが、今年初の潜りに行ってきた。
が、なんか耳抜きが上手く行かずに深く潜れなかったのが残念。小さめの魚が沢山群れていて海面近くでうだうだしてても楽しめたのは良かった。カメラは海中に持ち込まなかったので魚の写真はないw
アオリイカを追いかけまくって満足。井田は子供が多くてのんびりした感じだった。


案内表示板が謎。

ポケモン的にはコイルがよく出た気がする、あとビリリダマ(影のみ)。家ではまず見かけないのでコイルはゲット出来て満足。
尚、魚介は取ってはいけないがポケモンは禁止されてないようなので安心して捕獲作業できる。

02082016 pokemongo
しあわせタマゴをつかって進化させまくった結果66500ポイントを獲得し、Lv22になった。
次のレベルまであと90000ポイントなのはIngress並w
ジム見るとLv24とかいてわけわからん。カイリュー、カビゴン、ラブラス欲しいけど一体だけゲットしても強化出来ないからシャワーズ戦隊にしたほうが効率がいい気がする。
しあわせタマゴ+ポッポ進化のブースト使わなかったから3日くらいかかった。あと夏バテとポケバテでやる気のない日が1日ほどあったw
さらに、経験値よりは、いい感じのAR写真を撮る方に楽しみがシフトしているw

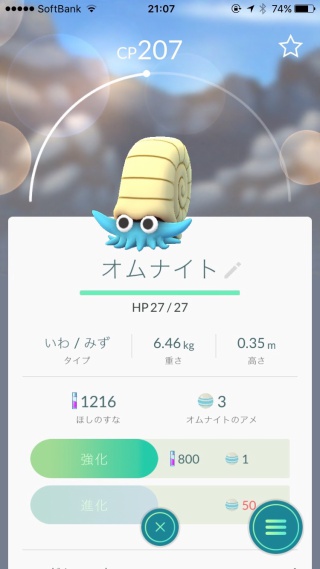
家にオムナイトが迷い込んできたので捕獲
ちょっと用があって静岡に行ってきた。駿府城でポケモン捕まえるぞーとか思っていたのだけど、暑すぎて断念したw
そもそも家をでる時点でこの富士山の状態がやばかった。静岡はやはり暑かった

久しぶりにコメヤスさんにでも行くかなーとぶらついたところ、美味しそうなラーメン屋さん発見。そして美味しかった。細麺の麺茹で35秒だったから、博多ラーメン的な麺なのに白濁した豚骨スープじゃなくてクドくない味でよい。替え玉もある。ここはつけ麺も美味しそうだった(麺は異なる)


用が済んだらまっすぐ帰る予定だったが、暑すぎて水分補給をせざるを得なかった。お茶エールを飲んだがあまりお茶感がなかった。


ところで、緑茶割りの緑茶はどのグレードの茶葉を使うのがいいのだろうか?
久しぶりに料理した。魚介のアヒージョと、鶏むねの塩レモンオーブン焼き。クスクスはサラダに使うよりもオーブン焼きの底に敷いたほうが好きかも。オーブン料理は入れっぱなしでいいのが楽なのでもう少しレパートリーを増やしたい。


コメヤスさんで19をゲット。酸が効いてて美味い。そして、日本酒が充実している居酒屋が富士にあるっていう話を聞いたんだけど、調べたら三島だった。今度行きたい。

28072016 pokemongo
IngressでいうところのLv8を達成した。大体一週間くらいか。

あるジムに挑戦するとGOって文字が大きくなるところでフリーズして強制終了、再起動で認証で5分くらい止まるというのを三回繰り返してイライラした。

あとは、ボール投げたら飛び出しちゃったからズリの実使おうとしたタイミングで雲隠れしちゃった場合フリーズするっていうのもバグかな。
ミニリュウゲットした。カイリュウまでの道は長いがのんびり育てるつもり。

27072016 pokemongo
60匹まとめて進化させるつもりだったのだけど…
夜、何故かカウントダウンするたまごが表示された。
それが「しあわせたまご」の効果だと気づいた時には既に10分以上経っていたという。(ポケットの中で勝手に使うことになってしまったようだ)
慌てて手元のポケモン進化させまくったのだけど、30000ポイントくらいにしかならなかったっぽくLv17->Lv18半分くらいまでしかあげられなかった。残りの分を息子と一緒に散歩してなんとか19にまであげて今日は終了。
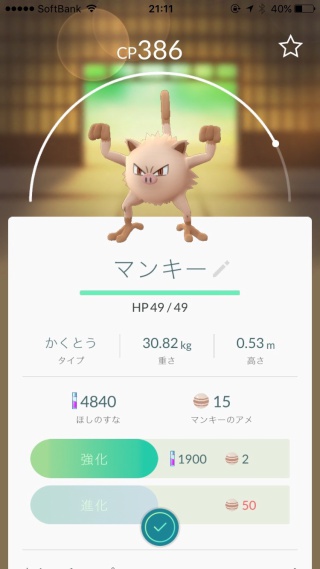
新しいポケモンは「カブト」「ゴース」「マンキー」
マンキーはキーキーうるさい息子にそっくりなので、息子の名前をニックネームにして育てることにしたw
それにしてもモンスターボールの枯渇は大問題だw 職場の周りにポケストップないから十分なボールを持って行かないと昼休みは孵化作業しかやれないのがツライw
23072016 pokemongo
昨日から始めてLV10にまで上げてみた。チームは青。

こんな感じでingressとは違ったまろやかさがあるから楽しいですね。

ただし、ジムにポコポコとポケモンのっけるだけでジム間の連携による面白さみたいなものがない。 ただ集めるだけだったらそのうち飽きるかなぁとしか思えない。バッジも基本的には集めたポケモンの数で決まる感じなので達成感は少ないかも。ingressみたいに勢力図の概念は欲しいなぁ。
尚、昨晩から近所のジムにLv24とLv22のプレーヤーがCP1400ゴダックとかCP1500ブースターとかCP1700シャワーズとかおいていて一体何なの?っていう… 運営の方でなんとかしてくんないかなぁ
22072016 ingress
前回のレベルアップからなんと10ヶ月…
前回レベルがあがってから、単にAPを稼ぐという作業がイヤになって離れていたのだけど、ウォーキングのために再開してから5ヶ月弱くらいかな。そしてトレッカーがプラチナになったので結構歩いたなぁと。トランスレーターもオニキスに行きそうなので続ければlevel16は見えているんだけどどうしたもんか(ウォーキングの延長としてもう少し頑張るかなぁ)
次のレベルもAP稼ぐだけだけど最近は家の周りは味方のポータルしかないからAP上げづらいんだよなぁ。L8バースターも400個くらい常にストックしてあるしなぁ…
といいつつポケモンGOを待ちわびているw