PKQuestというものがあるらしい
Programming Clojure
予約したまま5ヶ月近くたち、忘れかけてたProgramming Clojureが発送された。
Programming Clojure欲しいかも
この本ちょっと気になってきた
 Programming Clojure (Pragmatic Programmers)
Programming Clojure (Pragmatic Programmers)Stuart Halloway
Pragmatic Bookshelf / 2890円 ( 2009-06-03 )
どうなんだろうか?
21072008 java
インターフェース指向設計を読んだ
結合のこととか。デザインパターンとかRESTとか学んであればなかなか楽しく読めるとおもう。
1部2部は面白かったけど、3部の実例はいまいち興味がわかなかったので読み流した。あとなんというか、なんでかは言葉にできないんだけど、集中力を持続しつつ読みすすめにくい本だったなぁ(特に後半)
postitをちょこちょこ張っておいたので後でまた読み直すのでしょう。
bluecoveでBT-Mini2EDRが認識しない
jythonでwiiコントローラー使って遊ぼうかなと思いついたので朝から色々試してみた。
javaのライブラリ経由でアクセスすればよかろうと、WiiremoteJとbluecoveの組み合わせでいけるかなと思ったんだけどどうもBT-Mini2EDRを認識してない。
最初はWindowsで開発していたのですが、私の手持ちのBluetoothアダプタ(BT-Mini2EDRとBT-UD1)では何度やっても認識せず。しかたがなくmacbookにインストールすると全く問題なく接続できました。
どうも認識しないものらしい。これもまたMacBookを買えという見えない力が働いている証拠なのだろう。
Wiiでテルミンも面白そう。
jythonが2.2betaになってた
ここのところ更新がなかったので、もしかして終わっちゃったのかなと思っていたjythonが2.2betaになってた。
The Jython Projectから早速ダウンロードしてインストール。
これでpythonからjavaのライブラリが使えるが、ケモインフォ的にはCDKとdatasetをいじったりとか。あとRなんかで色々出来る気がする。
あと、QSAR用のレポジトリ欲しいナァ。論文に投稿するんだったら、sdfと活性値をsubmitしないと駄目みたいなPDBっぽい約束事とかあれば、いいと思うんだけど。再利用性が悪くてかなわん。
14012007 java
結城浩さんが新しいJava本を出すらしい
そういえば、去年の今頃も結城浩さんのJavaのデザパタ本読んでたなぁ。
で、今度はリファクタリングの本を出すそうです。
きれいなソースは読むのが楽で、修正するのが楽で、デバッグするのが楽です。これに対して、きたないソースはすべてが大変です。ではどうすれば「きれいなソース」になるのでしょう。きたないソースをどうすれば(新たなバグを出さずに)きれいなソースにできるのでしょう。それに対する試みの1つが「リファクタリング」です。
本書『Java言語で学ぶリファクタリング入門』では、 Java言語を使って、リファクタリングの本質をていねいに解説します。
かなり欲しげ。というわけで、書籍プレゼント企画に応募した。
Structure-CDKをperlで使う
smilesを描画するwebのインターフェースが欲しくて、色々作ってみた
必要なもの
- Java
- Structure-CDK (include cdk)
- opsin-big-0.1.0
- Perl
- Inline::Java
-
Chemistry::OpenBabel( need for iupac2png)
-
http://depth-first.com/articles/2006/10/17/from-iupac-nomenclature-to-2-d-structures-with-opsin
- http://depth-first.com/articles/2006/08/28/drawing-2-d-structures-with-structure-cdk
- http://sourceforge.net/projects/structure
ここら辺を参考に書いた。できたものはCDK-Util-0.01.tar.gzにおいておいた。
- smi2mol
- smi2png
- IUPAC2CML
が可能。IUPAC2PNGも書いたのだけどmake testで豪快にこけるので無視してインストールしてください。podの細かいとこも気にしないように。Iupac2Pngはcdkでcmlをmolに変換するあたりがわからなくてあきらめたけど、そこのコードの部分だけ手直しすれば動くと思う。
っていうか、iupac2pngはopenbabelのモジュール使えばいいじゃんって気付いた途端もういいやってなってしまった。書式コンバートはopenbabelのほうがわかりやすい。ちょっとコード量は増えるけど、こんな感じ。
use strict; use warnings; use CDK::Smi2Png; use CDK::Iupac2Cml; use Chemistry::OpenBabel; my $iupac_name = shift; my $ic = CDK::Iupac2Cml->new(); my $cml = $ic->convert($iupac_name); my $obMol = new Chemistry::OpenBabel::OBMol; my $obConversion = new Chemistry::OpenBabel::OBConversion; $obConversion->SetInAndOutFormats("cml", "smi"); $obConversion->ReadString($obMol, $cml); my $smi = $obConversion->WriteString($obMol) or die "$! not converted"; $smi = (split(/\t/, $smi))[0]; my $smic = CDK::Smi2Png->new(); $smic->writePNG($smi,200,200,'/home/kzfm/test.png');
で実行する。Imatinibくらいは認識するようだ。
./iupac2png.pl "4-[(4-methylpiperazin-1-yl)methyl] \ -N-[4-methyl-3-[(4-pyridin-3-ylpyrimidin-2-yl)amino] \ -phenyl]-benzamide"
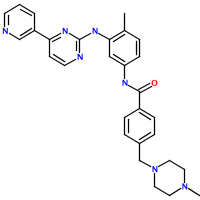
あとはcatalystとsmi2pngを組み合わせて、chemichoのwebインターフェースに使ったり、iupac2pngでcgiでもつくってbookmarkletでポストできるようにしとけばpubmedとかのIUPAC名をいつでもひける。辞書サーチのプラグインで右クリック呼び出しでもいいけど。
18102006 chemoinformatics java cdk
CDKで型があわねーとかいうエラー
Depth-FirstでみっけたStructure-CDKが使いたいということでcdk-20050826.jarからcdk-20060714.jarにあげたらこの前つくったモジュールが動かなくなった。
sdg.setMolecule(mol); sdg.generateCoordinates(); mol = sdg.getMolecule();
のところで、
Smi2Mol_899b.java:49: 互換性のない型 検出値 : org.openscience.cdk.interfaces.IMolecule 期待値 : org.openscience.cdk.Molecule mol = sdg.getMolecule(); ^ エラー 1 個
もげー、型が違うってなんでじゃ?

 インターフェイス指向設計 アジャイル手法によるオブジェクト指向設計の実践
インターフェイス指向設計 アジャイル手法によるオブジェクト指向設計の実践 増補改訂版Java言語で学ぶデザインパターン入門
増補改訂版Java言語で学ぶデザインパターン入門