18082009 chemoinformatics bioinformatics q-chem
ドッキングシミュレーション関係の本だろうか。
あとGaussian
Gaussianは使ってないのでこっちはあまり興味をそそられないが。
18082009 chemoinformatics bioinformatics q-chem
ドッキングシミュレーション関係の本だろうか。
あとGaussian
Gaussianは使ってないのでこっちはあまり興味をそそられないが。
13082009 chemoinformatics R PRML
20072009 chemoinformatics bioinformatics macbook
trunk(Rev: 3829)をDLしてきてコンパイル
svn co https://pymol.svn.sourceforge.net/svnroot/pymol/trunk pymol
freetypeがらみのヘッダーがないとかいうエラーがでてきたので
cd /usr/X11R6/include sudo ln -s freetype2/freetype freetype
とsymbolic link張って解決
 Bioinformatics Programming Using Python
Bioinformatics Programming Using Python18072009 chemoinformatics bioinformatics
07072009 chemoinformatics work
昨日、ワークショップで雑談してて、メディシナルケミストリーにおけるフッ素の役割がとか。
ハロゲンのくせに、Cl,Brとはちょっと違うよねとか。
 Bioorganic and Medicinal Chemistry of Fluorine
Bioorganic and Medicinal Chemistry of Fluorine06072009 chemoinformatics work
DMPK,TKを考慮したドラッグデザインの際に考慮することなど。
多分メディシナルケミスト向けなんだろうなと思うんだが、コンパクトにまとまっているので、chemoinformaticsのヒトが読んでも参考になることが多い。
 Pharmacokinetics and Metabolism in Drug Design (Methods and Principles in Medicinal Chemistry)
Pharmacokinetics and Metabolism in Drug Design (Methods and Principles in Medicinal Chemistry)薄いわりにはちとお高めだが、投資と思い割り切った(今回は)。
30062009 chemoinformatics
Avogadroだとタンパク質のアミノ酸とかを簡単にエディットできないな。
スクリプト書いてリガンドから半径なんぼ以内の残基を切り出すようにすればさくさく動くかも。
24052009 chemoinformatics work
リードホッピングとはつまるところ、選択されなかったより良い未来を、過去にさかのぼり選択しなおすことだと思うのだ。または、現在の結果が(成功にせよ失敗にせよ)選択されたことで、過去の時点の確率が変化したというベイズっぽい解釈でもよいかと。
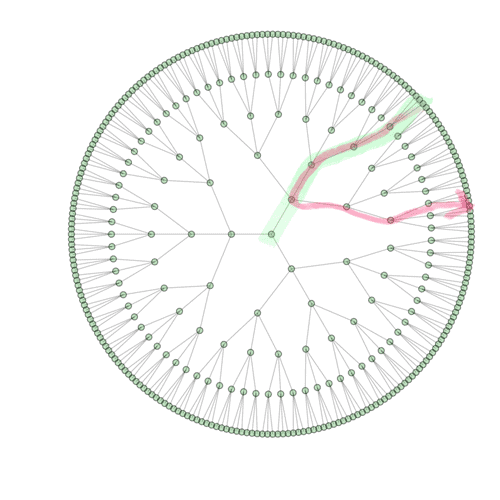
上の図でいうと外側のサークルを類似性とかで辿ったとしてもホッピングなんて成し遂げられなくて、もっと、最初の化合物に近いところにさかのぼりつつうまい分岐点で辿りなおさないといけない。
そのためには化合物のデータベースがここの化合物がどう継承されるかを記録されプロジェクト毎にツリーとして表せるようにしておかないといけないんだろうなぁ、なんて思うわけだ。
22052009 chemoinformatics
12052009 chemoinformatics DMPK
はじめて読むならこれがお薦めか。ケモインフォマティクスな僕にとってはある程度数式とか統計の話がくっついていたほうが、知識と照らし合わせながら覚えていけるので分かりやすかった。
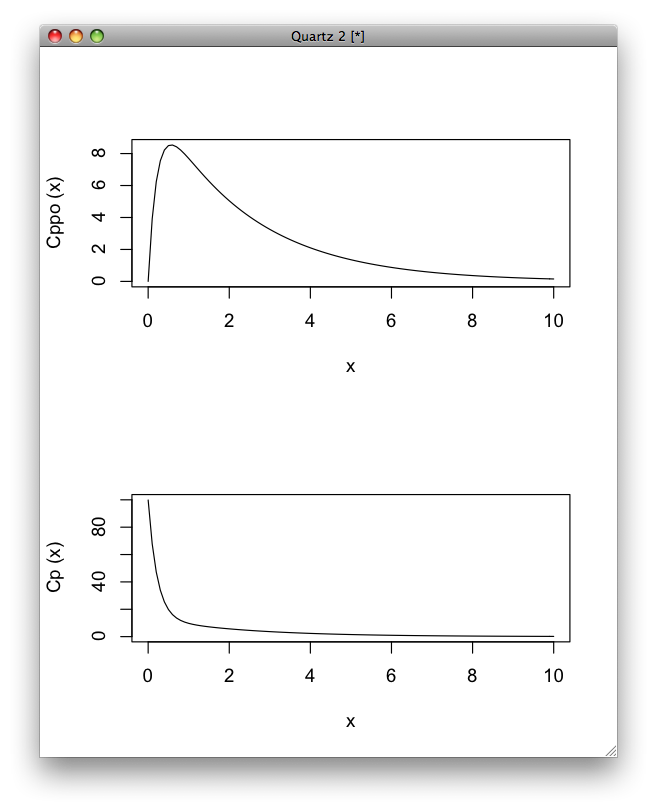
ちょっと7章だけプロットしてみた。
Cp <- function(t){86.22 * exp(-4.55 * t) + 13.63 * exp(-0.438 * t)
Cppo <- function(t){456700/(9128*(4.567-0.438)) * (exp(-0.438 * t) - exp(-4.567 * t))}
par(mfrow = c(2,1))
plot(Cppo,0,10)
plot(Cp,0,10)
演習の多そうなこっちもやっとくべきか。