朝からRをいじりたくなって、「あーそういえば2.8出たんだっけ」とか言いながら、ソース落としてきてmacbookでコンパイルをしてみたがこけた。まぁ、2.7.1でRpy動くのでとりあえずいまのところはそっとしておいてやろうということで負けを認めない方向で。
で、RandomForestは好きな時に使えるようになっておかないといけないなぁといつもお世話になっている連載を読み直した。というより、いままで上っ面を押さえただけでわかった気になっていただけだったので、ちゃんと読んだ。
- 与えられたデータセットからN組のブートストラップサンプルを作成する。
- 各々のブートストラップサンプルデータを用いて未剪定の最大の決定・回帰木を作成する。ただし、分岐のノードはランダムサンプリングされた変数の中の最善のものを用いる。
- 全ての結果を統合・組み合わせ(回帰の問題では平均、分類の問題では多数決)、新しい予測・分類器を構築する。
BaggingとRFの大きい違いは、Baggingは全ての変数用いるが、RFでは変数をランダムサンプリングしたサブセットを用いることができるので、高次元データの計算が容易であるである。ランダムサンプリングする変数の数Mはユーザが自由に設定することができる。Breimanは、Mは変数の数の正の平方根をと取ることを勧めている。
あとはこっちも参考になった。
RandomForests とは?
Random Forests では、個々のコンポーネントモデルを生成するのに全く新しい方法を利用する点が異なります。逆説的ですが、「強い」独立モデル同士では、モデルが全体として高いパフォーマンスを引き出すよう有効に結合しません。むしろ「弱い」独立モデル同士の方が効果的な結果をもたらす傾向があるのです。この理由として挙げられるのは次の 2点です:
まずひとつは、複数のモデルを組合せたり平均化を行っても、個々のモデルがお互いに非常に類似している場合には際立った精度の改善が見られないという点があります。極端な場合、各モデルが全く同一のコピーだとすると、平均化することによってそこから得られる利益(ベネフィット)は全く何もありません。組合せ技術で何らかの利益をもたらすためには、組合せるモデル同士はお互いに異なっている必要がある訳です。
要するに予測精度はある程度弱くてもよいからできるだけ直交するようなモデルを組み合わせることが大切ということか。
Rで使う
CRANにrandomForestというパッケージがあるのでそれを利用する。unsupervisedで遊んでみた。
> library("randomForest")
randomForest 4.5-28
Type rfNews() to see new features/changes/bug fixes.
> data("iris")
> set.seed(17)
> iris.urf <- randomForest(iris[, -5])
> MDSplot(iris.urf, iris$Species)
### plot ###
> importance(iris.urf)
MeanDecreaseGini
Sepal.Length 17.33820
Sepal.Width 15.19356
Petal.Length 17.30337
Petal.Width 12.92124
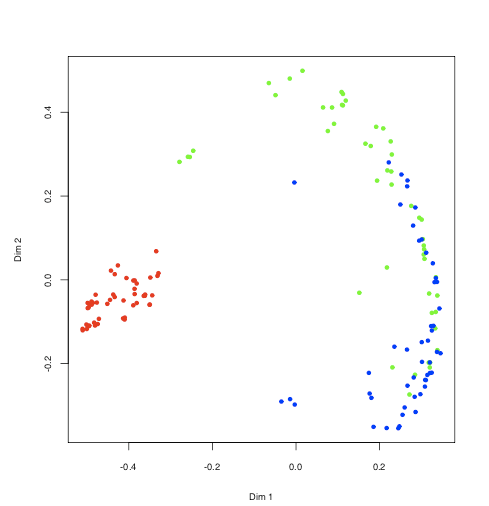
こっちはsupervised
> data(iris)
> iris.rf <- randomForest(Species ~ ., iris, proximity=TRUE,
+ keep.forest=FALSE)
> MDSplot(iris.rf, iris$Species)
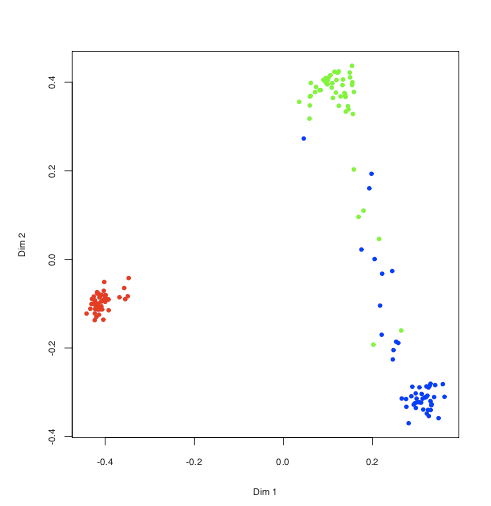
他にもimportanceとかgetTreeといった関数が重要そう。

 Rで学ぶクラスタ解析
Rで学ぶクラスタ解析

 Rによる医療統計学
Rによる医療統計学 プログラミングのための線形代数
プログラミングのための線形代数 Rの基礎とプログラミング技法
Rの基礎とプログラミング技法 Mastering Algorithms With Perl
Mastering Algorithms With Perl