23022019 life
システムバイオロジー関連のセミナーに参加したので帰りは神田の大松に寄ってイワシってきました。
ウドとポテサラ


ワンタン揚げと〆イワシ


なめろうとレモンハイ


美味かった。
23022019 life
システムバイオロジー関連のセミナーに参加したので帰りは神田の大松に寄ってイワシってきました。
ウドとポテサラ


ワンタン揚げと〆イワシ


なめろうとレモンハイ


美味かった。
23022019 bioinformatics
読み終わった。
wetのバイオロジーやってるけどIT側も使いこなせるようになりたいひとが読むべき本かなと思いました。まぁうちのバイオインフォのチームは必読でしょう。
私はThe OBO Foundryを知ることができたので満足。
今年はバイオインフォの活動も頑張りまっす。
19022019 chemoinformatics q-chem RDKit Psi4 Psikit
Considering the conformational effects of the compound is important in Structure Based Drug Design, this paper discussed about it, in terms Protein-Ligand binding using torsional scan of each ligands(PDB:2JH0,2JH5,2JH6). They calculated torsional energy, and explained the relation between inhibitory activity and torsional energy.
Torsional scanning is the task of the quantum chemistry rather than that of chemoinformatics. But I wanted to conduct quantum chemical calculation as an extention of chemoinformatics way, so I implemented it in Psikit
jupyterでRDKitからのB3LYP/6-31G*でのDFT計算さっくりうごくの素晴らしい。
15022019 FMO
面白そうなお題が与えられたのでやってみた。ちなみにすぐにFMO計算できる環境なので面白そうな複合体の情報があれば教えてください、計算して可能な範囲で公開します。
これFMO計算でどれくらい定量的に説明できますかね?@fmkz___ https://t.co/iMN2Kt1QHu
— torusengoku (@torusengoku) 2019年1月31日
6FS6のPIEDA。グラフの見方は縦軸がエネルギーで安定な相互作用だと負にでます。EXは反発項なので常に制に出ます。横軸は残基で本来はN末から順番に並んでいますが、みやすさのために相互作用が5kcal/mol以上の相互作用のみ表示するようにしています。
見て分かる通りLYS034, ILE038との相互作用が強い。ILEとのDIがかなり強いことがわかる。8kcalあるし相当強いですね。 尚、EXちょっと強めに出てるのはエナジー最適化ちょっとまずってるのか、立体的に込み入った分子だから力場が良くなかったのかも。
で、これをI38Tにして差分を取ると38番目の残基のDIが2.5kcal/molくらい不安定になってEXが1kcal/molくらい安定化しました。それ以外の大きな差はみられなかった。今回EXに関しては大目に見るとして、経験的にはDI項結構効いてるんじゃないですかね?koff測定すればもっとはっきりしたこと言えるかなぁと思いました。
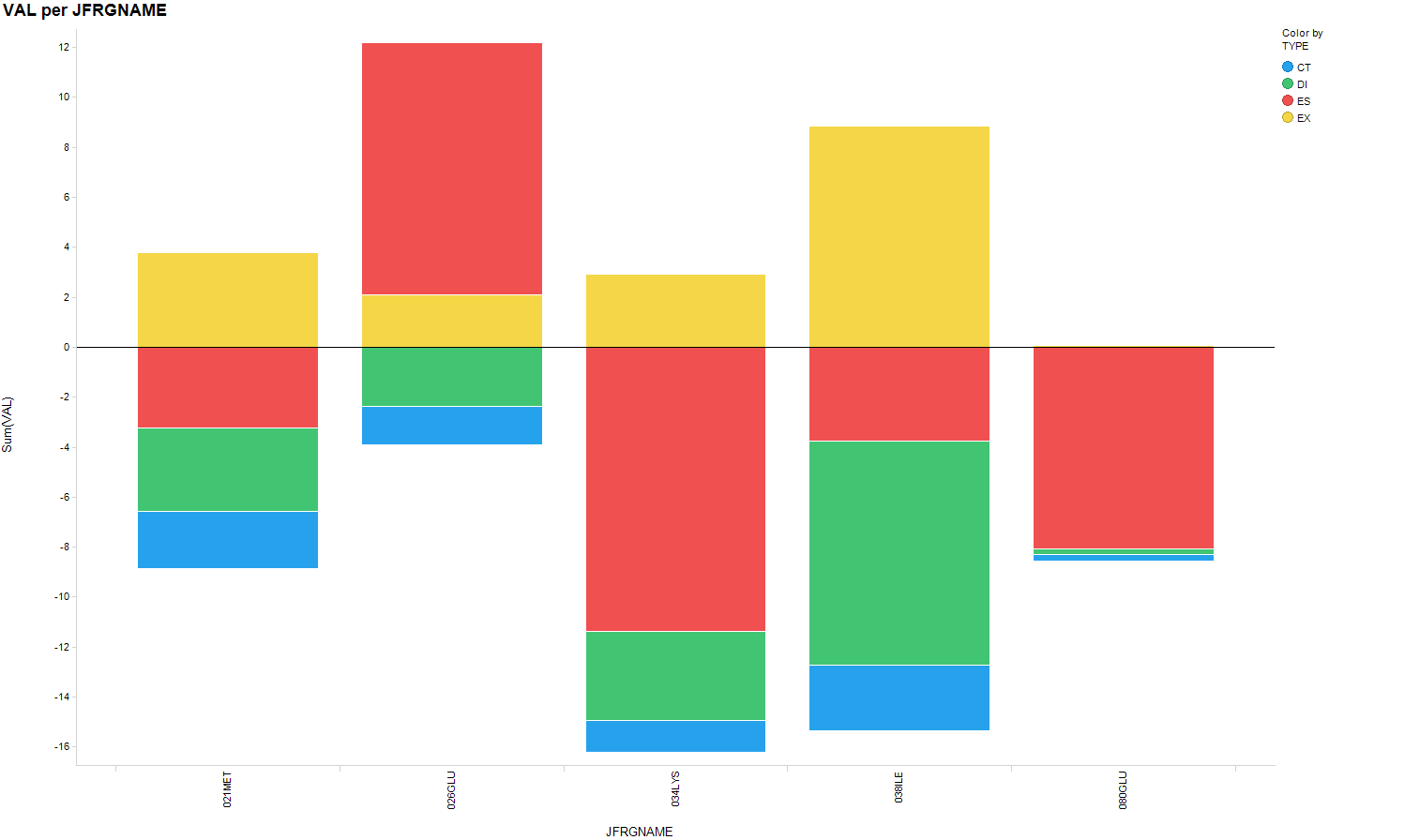
上のIFIEを実際にリガンド結合サイトで塗ったものです。青いところが安定な相互作用しているところで赤いと反発してます。
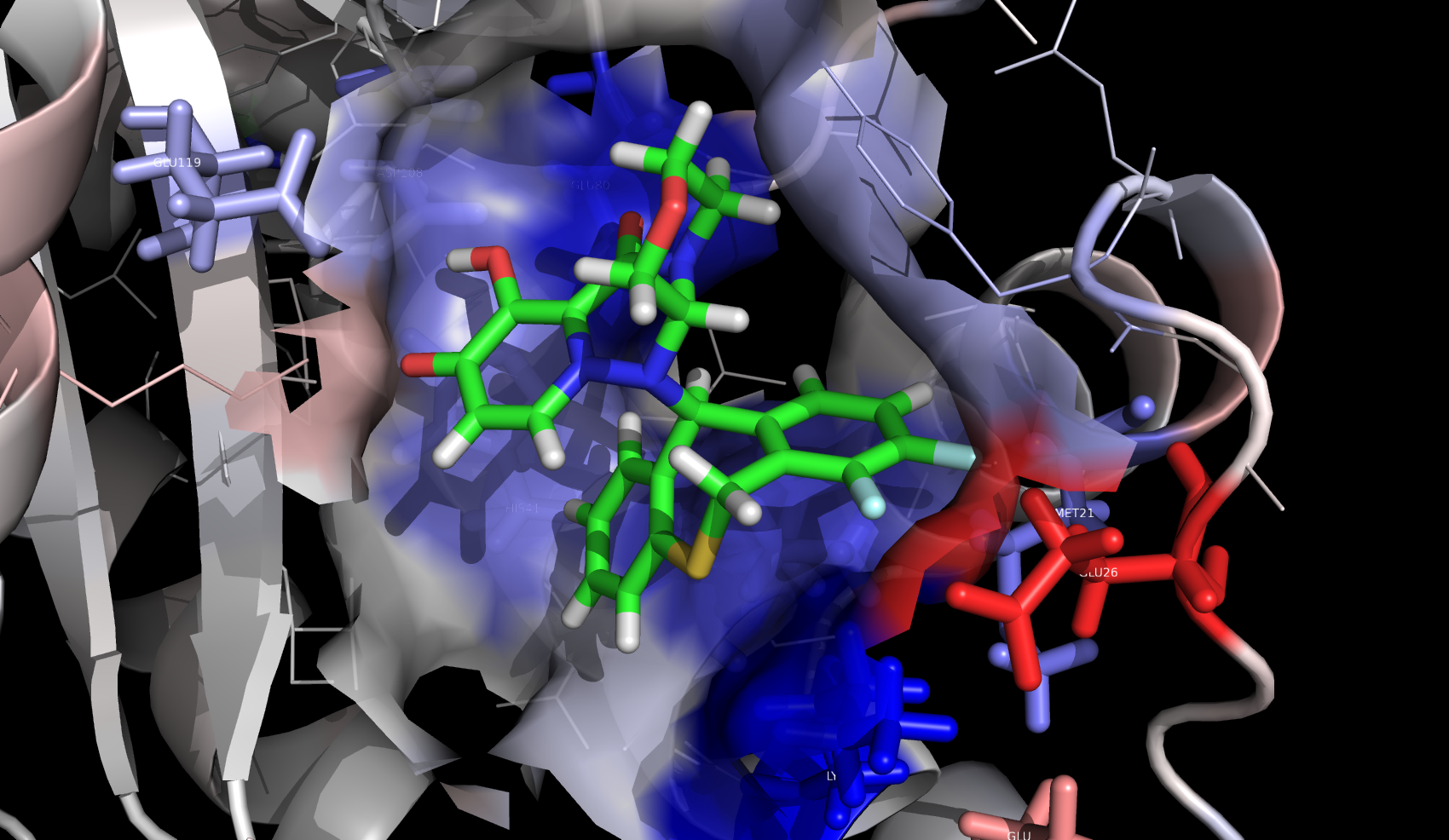
DIだけの成分で塗り直したものです。ILEとこんなに強いDIで安定化してる例見たことないから結構感動した。粘着感がありますねw
DIで安定化していてSARと突き合わせて見られる例としてはFXaのS4ポケットがあります。FXaは複合体結晶構造もSARの論文もたくさん出ているので興味があれば調べてみるとよいと思います。
11022019 bioinformatics
先週のMishima.sykの懇親会では著者による「サイン付き著者割生命科学データ解析を支える情報技術」が人気を博しておりましたが、うっかり速攻アマゾンで予約するという失態を犯した私のもとにも無事に届きました。
私は今月からバイオインフォマティクスの部署も担当することになったので、もし 「企業で俺のバイオインフォマティクス力の高まりを顕現させてみようかな?」 という方がおられる、または周りにおられる、もしくは共同研究してみてもいいんじゃない?みたいな研究室があればお知らせいただければ、美味しいクラフトビールなど飲みながら詳細詰めさせていただければと思っていますので右のアイコンからコンタクトよろしくおねがいします。興味のある仕事は勉強がてら今後エントリにあげようと思っています。
今回、これ最高だったので資料が上がって嬉しい。
追いかけないといけないことが増えた。
10022019 chemoinformatics
今回バイオインフォマティクス寄りの発表がいくつかあったので、非常に勉強になった。テンソル分解ちゃんと勉強しないとなと思いました。
ハンズオンのほうは、condaはあまり万能じゃないなと。油断してた。Windowsだとunzipのcondaバイナリないとか、SMILESを""で囲うとだめだとかよくわからないエラーに悩まされた。まぁでもWindowsでもREINVENTが動いたので良かったとしよう。
お昼は王味で小エビラーメン

懇親会の刺し身

二次会のリパブリューのラムチョップ

そしてアルコール度数17.5%のビール

次回もまた楽しくやりましょう
01022019 chemoinformatics
参加はこちらから。
今回の会の趣旨とか狙いみたいなものはこちらを御覧ください。
私はSitagliptinのアナログを出すようなジェネレータを動かすハンズオンをやろうかなと考えています。デモのために訓練300回させたモデルでとりあえずjupyter notebook作ってみましたが、出来がイマイチなので週末にでも3000 iterationくらい回したモデルを作り直して更新する予定です。
12012019 life
One of the founders of Mishima.syk passed away.
We can fly But We want his wings We can shine even in the darkness But We crave the light that he brings Revel in the songs that he sings My angel XXX
03012019 chemoinformatics
あけましておめでとうございます、今年もよろしくおねがいします。
昨日まで日本酒飲みまくってグダグダしていたので、今日からそろそろ始動しようかなと思います。@iwatobipenがアクティブにGitHubにプッシュしまくっているみたいなので、Mishima.sykのタイムスケジュールも更新しましたが、あと40分くらい空いているのでLT入れたい人は早めに時間を確保してください。
今回はGenerator mini hands-onをやることにしました。理由は、第404回CBI学会講演会ではThierry KogejさんにMolecular de novo design through deep learningと似たような公演をしてもらう予定にしているからです。
スライド3でDe novo molecular generation with deep learning has developed very rapidlyってあるんだけど、今大体どのくらいの化合物生成してくるのかを知らないと、公演楽しめないと思うんですよね。この手の話って知らないで聞くと期待感あふれるストーリーなのか、現実味を帯びているのかいまいち掴めないという。
というわけで彼らの提案しているREINVENTを実際に動かして、化合物提案がどんなもんなのかを見てみようというハンズオンになります。予めChEMBLでトレーニング済みのモデルはこちらで用意しましたのでjupyterでちょっとコード打てば動くようにする予定です。
本当は参加者に、それぞれ上市された薬剤のもとの特許を渡して、生成モデルがどのくらい生成したら実施例をカバーすんのかな?っていうのをやってもらうハンズオンにしたかったんだけど、それはGPU必須っぽいので今回は断念しました。
参加はこちらから。
ハンズオンをやってから改めて読むと味わい深いエントリはこちらです。
今年を振り返ると書いたエントリ数は100弱だった。昔は1エントリ/day以上は書いていたので最近はブログに対する意欲は薄れているが、後から振り返るためにもう少し書くようにしたい。
今の会社に入って2年ちょいだけど、今年は成果が沢山でてよかったと思う。チームで社外発表を全部で10以上できたし、来年講演に呼ばれているの確定なの4,5はあるしね。来年も社外発表を通して議論、フィードバックをもらいながら成長していきたい。
または overwhelming growth ってやつ?w
一方で去年の振り返りに埋め込んだTEDの動画にあるように、去年はタイミングが良かったというのもあると思うんですよね。「半歩先の仕事の発表だったからウケた」みたいな。サービスとしてはありなんだろうけど、サイエンスとしてはどうなのかなーと思わなくもない。マジョリティが理解できる程度にしか先に進んでないってことだから、すぐに追いつかれる強みにしかならんのかなーとも思うので、そのあたりも考えていかねばいけませんね。と、そういうことを考えられるようになったのも進捗した結果なのでそれはそれで嬉しいことですが。
来年は 「単純なAI based XX」っていうネタも落ち着いて、ドメイン知識が必要ななにか新しいことをプラスしないと評価されなくなってくるっていうようになるんじゃないかなぁーって思います。特にケミストリーはAIっていう焼畑農業である程度焼き尽くしちゃった感があるしね。そのあたり、少し進めていければいいなと思っています。
あと来年はちょっとBioinformaticsのお手伝いもしないといけないのでそっちもきちんと成果を出せるようにしたいけど。まずは環境を整えないといけないなって感じですね。あとバイオインフォの情報も追いかけないといけないので大変になりそう。
あと英語力をもっとあげていきたい。僕もエントリ英語にしようかな。
手術というものを初めて経験した、盲腸だけど。それからPokemon GoのトレーナーレベルがMax(40)に到達したら、あまり運動をしなくなってしまった。良くないのでなにかしないとなーと思っている。