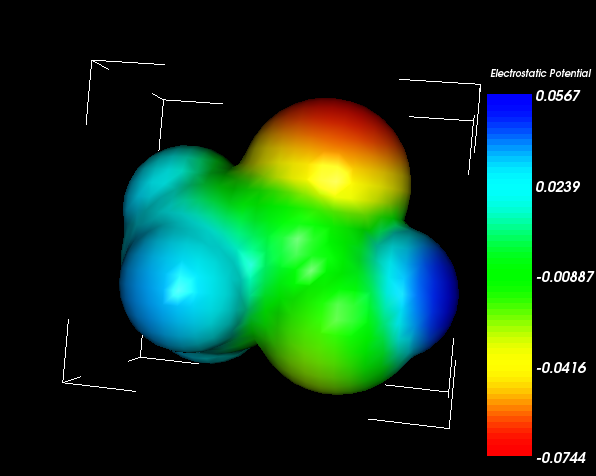
Cation-πってどのくらい安定化すんだろうか?と思っていたので計算してみる。
RCSBを適当にあさって見つけた、1R9LのBETとTRP188を切り出してモデル化する。
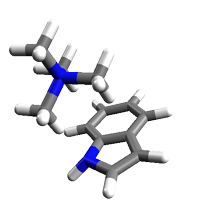
で、GAMESSで計算。
最初は6-31G+(d,p)で最適化しようと思ったのだけど、macbookだと全然終わらん。なのでSTO3Gでいいやと。TRPは無駄に計算量が増えるのだけどPHE,TYRあたりでモデル化できるのであればmacbookでも6-31G+(d,p)で計算できるような気がする。
------------------------------------
RESULTS OF KITAURA-MOROKUMA ANALYSIS
------------------------------------
HARTREE KCAL/MOLE
ELECTROSTATIC ENERGY ES= -0.011414 -7.16
EXCHANGE REPULSION ENERGY EX= 0.008788 5.51
POLARIZATION ENERGY PL= -0.001679 -1.05
CHARGE TRANSFER ENERGY CT= -0.007053 -4.43
HIGH ORDER COUPLING ENERGY MIX= 0.000166 0.10
TOTAL INTERACTION ENERGY, DELTA-E= -0.011193 -7.02
水素結合と同じくらいの強さかな。
以下、STO-3Gで構造最適化した座標を使ってつくったインプット
$BASIS GBASIS=STO NGAUSS=3 NDFUNC=1 $END
$CONTRL SCFTYP=RHF RUNTYP=MOROKUMA MAXIT=200 ICHARG=1 MULT=1 $END
$MOROKM IATM(1)=17 ICHM(1)=1 $END
$DATA
W188_BET.out
C1
N 7.0 26.56861 22.88472 72.47206
C 6.0 25.05591 23.05763 72.38527
C 6.0 27.18276 23.04159 71.08205
C 6.0 27.14794 23.94295 73.40648
C 6.0 26.89124 21.49601 73.01604
H 1.0 24.65761 22.29324 71.71973
H 1.0 24.84300 24.05067 71.99229
H 1.0 26.95089 24.04166 70.71972
H 1.0 26.73892 22.29197 70.42917
H 1.0 28.26387 22.89551 71.15590
H 1.0 26.89847 24.92550 73.00913
H 1.0 28.22884 23.81270 73.44995
H 1.0 26.70726 23.81001 74.39332
H 1.0 26.46855 20.75492 72.33939
H 1.0 26.44825 21.40230 74.00640
H 1.0 27.97398 21.38820 73.06968
H 1.0 24.63622 22.94695 73.38408
C 6.0 30.61324 20.49644 72.40614
C 6.0 30.47183 21.03406 71.19420
C 6.0 30.79531 21.59051 73.36029
C 6.0 30.75216 22.77934 72.62888
C 6.0 31.00908 21.63948 74.74382
N 7.0 30.43745 22.45766 71.27884
C 6.0 30.92671 24.02761 73.23103
C 6.0 31.18435 22.86580 75.34238
C 6.0 31.14962 24.05129 74.58925
H 1.0 30.33993 20.55500 70.23268
H 1.0 31.04481 20.72825 75.32842
H 1.0 30.86231 23.00634 70.52231
H 1.0 30.90815 24.93911 72.64846
H 1.0 31.36065 22.92555 76.40870
H 1.0 31.30378 24.99933 75.08996
H 1.0 30.61446 19.44515 72.64676
$END

 タンパク質計算科学 ―基礎と創薬への応用― [CD-ROM付]
タンパク質計算科学 ―基礎と創薬への応用― [CD-ROM付] Gaussianプログラムによる量子化学計算マニュアル 計算入力法から実験値との比較まで
Gaussianプログラムによる量子化学計算マニュアル 計算入力法から実験値との比較まで
 すぐできる 量子化学計算ビギナーズマニュアル (KS化学専門書)
すぐできる 量子化学計算ビギナーズマニュアル (KS化学専門書) The Fragment Molecular Orbital Method: Practical Applications to Large Mokecular Systems
The Fragment Molecular Orbital Method: Practical Applications to Large Mokecular Systems 基礎量子化学―軌道概念で化学を考える
基礎量子化学―軌道概念で化学を考える 有機化学のための分子間力入門
有機化学のための分子間力入門