04122007 Catalyst chemoinformatics perl bioinformatics
Catalyst + Open Flash Chart: Fancy graphs with minimal fuss
こんなのが10分程度で作れてすごいいい感じ。昔作った遺伝子発現のやつをこっちにかえるといいかもしれん。
ちょっとしたもの作るにはChart::OFCもいいですな。
04122007 Catalyst chemoinformatics perl bioinformatics
Catalyst + Open Flash Chart: Fancy graphs with minimal fuss

こんなのが10分程度で作れてすごいいい感じ。昔作った遺伝子発現のやつをこっちにかえるといいかもしれん。
ちょっとしたもの作るにはChart::OFCもいいですな。
08102007 chemoinformatics Meadow bioinformatics HOP
05102007 bioinformatics
29092007 chemoinformatics bioinformatics
16092007 bioinformatics
おー、おもしろそうだ。
バイオインフォではゲノムからメタボロミクスまで幅広いデータを扱いますが、これらの大規模なデータを理解するためには適切なビジュアライゼーションと使いやすいユーザーインターフェイスが欠かせません。 そのような状況の中で、ネットワークの可視化を行う Cytoscape や、複雑な系のシミュレーション結果を可視化する E-Cell 3D など魅力的なアプリケーションが開発されてきています。また、Web2.0 時代を迎え AJAX などの技術を活用した効果的なインターフェイスも普及してきました。そこで、今回は「ビジュアライゼーション」にフォーカスを絞った研究会を開催したいと思います。
データの量と種類が増えたときに目という(中途半端な)デバイスにどうやって認識させるかというのはかなり興味がある。
ケモインフォだとなかなかSAR表から先にいかないのが不思議で仕方ない。多次元最適化とかいってexcelの表がZ列超えんのが普通なのに、、、よく読めるなと感心する。
あとbioflashとかどうなってるんだろうか?あれもAjaxよりも視覚にうったえる力が強いと思うので面白そうだなと思ってたんだけど。
12082007 chemoinformatics perl bioinformatics
さて、bioinformaticsとかchemoinformaticsとか言われてるようなアレは、化学とか生物学に対して情報学的観点からアプローチしたりするわけです。こういう点から見ると、薬ってもんは蛋白質にうまいことはまる鍵みたいなもんで、蛋白質の穴にいい感じではまるような化合物を(コンピュータを駆使して)設計していくのが(コンピュテーショナルな)ドラッグデザイン(CADD)という分野だヨ。
で、蛋白質というものは複数のアミノ酸から構成され(100から数百)そしてアミノ酸は20種類程度存在し、それぞれ数十の原子から構成されているわけだ。要するに一対多の階層構造をとる。
protein -> amino-acid -> atom
みたいな。
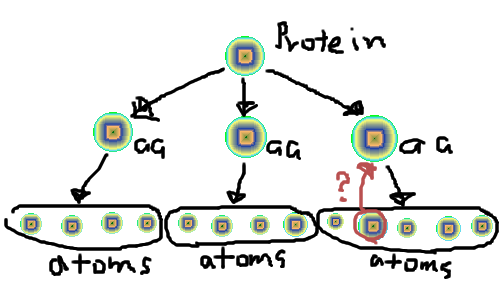
でそれぞれprotein aminoacid(aa) atomみたいなクラスを用意すれば
$atom1 = Atom->new({name => 'C1', type => 'C'});
...
$aa1 = AminoAcid->new({name => 'GLY', atoms => [$atom1, $atom2, ]})
...
$protein = Protein->new({name=> 'ProteinA', aminoacids => [$aa1,$aa2,$aa3...]});
みたいにそれぞれ配列に突っ込めば蛋白質を表現できて、あるatomオブジェクトを与えられた場合にそれがどのアミノ酸に属しているのか調べるのに
for my $aminoacid ($protein->aminoacids){
for my $atom ($aminoacid->atoms){
return $aminoacid if $qatom == $atom;
}
}
みたいにdepthfirstで探索していけばいいんだろうけど、ちょっと探索効率が悪いので、atomオブジェクトに $atom->{parent} = $parent_aminoacidみたいに親のアミノ酸オブジェクト返すような属性追加したんだけど、これだと構造が複雑になってなんか気持ち悪い。
他にうまいやり方ってあんのかなと思ったお盆の夏2007。
06082007 chemoinformatics R bioinformatics
LLで話されるような内容とは対極に近い、Rの統計解析環境としての本。かなりニッチなところをついてきた感があるが、Rのユーザー層を考えるとこっちのほうが普通の使い方なのかも。
RにおけるR Commanderってelispに対するemacsみたいな感じなのかな。
あとRってpythonみたいに対話環境がデフォルトだし。
なんつうか不思議な言語ですな。
08042007 bioinformatics
PharmGKBのGoogle TechTalks、結構意欲的というか面白い。
Web ServiceAPIがあるので、うまく使うといいかも。
03042007 bioinformatics
偶然みつけた。
Perl: Doing something useful with the secrets of life
おー懐かしい。
でも、Structural Bioinformaticsはバイオロジーとかインフォマティクスのレイヤーからでなくて化学からアプローチしていくほうがおもろいねというか個人的に好き。
31032007 chemoinformatics bioinformatics pymol
Wiiリモコンを使ってPyMOLをLet's Note R4上で動かしてみたのでXactiで撮ってみた。
Bluetooth USBアダプタは安いやつってことでBT-Mini2EDRを選択。
パソコンにリモコンを認識させたら、WiinRemoteでWiiリモコンをマウスとして操作。
結局、傾きとボタンの組み合わせで回転したりズームしたりしてるだけだけなので、思い通りに動かすのにちょっと苦労するんだけど、出来合いのものをチョロッと動かして見せるようなプレゼンのときにはいいかもしれん。
今のところマウスの方が圧倒的に操作性がいいんだけど、センサーバーを使ってきちんとイベントをアサインすれば、Wiiで遊んでいるようなような操作感が得られて、いい感じのデバイスになるような気はする。
こういうデバイスが使えるようになるとViewerのほうも調子のいいものが求められるようになっていく気がする。
例えば、遠くから眺めてるときは、リガンド-サーフェース(タンパク質)として表現されていて、ズームしていってアクティブサイト近くになるとAtom-Atomのスティック表現になったりとか、さらに、置換基-アミノ酸のレベルまでズームすると軌道とかで表現したりとかシームレスに切り替わる(google mapsみたいに)ような。
そんなViewer欲しい。