去年からダラダラと作っていたPythonのGAMESSラッパーをPyPIにあげてみたけど、初めてなので、なんかおかしい部分があったら指摘してもらえるとありがたいです。
登録は以下のサイトを参考にした
実際にやってみて分かりづらかったところはやっぱライセンスかなぁ。逆にclassifierのところはlist見ながら該当する部分をコピペしてけばいいだけだったので思っていたよりは楽だったな。
去年からダラダラと作っていたPythonのGAMESSラッパーをPyPIにあげてみたけど、初めてなので、なんかおかしい部分があったら指摘してもらえるとありがたいです。
登録は以下のサイトを参考にした
実際にやってみて分かりづらかったところはやっぱライセンスかなぁ。逆にclassifierのところはlist見ながら該当する部分をコピペしてけばいいだけだったので思っていたよりは楽だったな。
23062011 Python
最近またまたエキスパートpythonを読みなおしていてnosetests --with-doctest便利だわってことになった。何周読むねんって感じなんだけどちょこちょことつまみながら順調に吸収しております。ほんま良書やと思います。
で、rstでdocテストすんだったら文書管理はSphinxでいいじゃんと思って調べたらFlaskがそうなってた。
$paster create ... $tree . ├── myapp │ └── __init__.py ├── myapp.egg-info │ ├── PKG-INFO │ ├── SOURCES.txt │ ├── dependency_links.txt │ ├── entry_points.txt │ ├── not-zip-safe │ └── top_level.txt ├── setup.cfg └── setup.py 2 directories, 9 files
pasterで雛形作ってdocsディレクトリ掘って移動してsphinx-quickstart
$mkdir docs $cd docs $sphinx-quickstart ...
これで雛形が出来上がるのでまずはSphinxのドキュメントを書きながらどういうAPIにしたいのか練って実装していく。
そしておもむろに
nosetests --with-doctest --doctest-extension=.rst
これでOKな気がするんだけどもっとイイやり方あるんだろうか?
22062011 chemoinformatics Python openbabel GAMESS
去年書いたGAMESSラッパーに手を加えてGitHubにあげた。ヘッダーの生成まわりはもっとやらないといけないんだけど、基底関数とコントロールまわりは動くようにした。といっても一点計算と最適化ぐらいしかしないんだけど。
こんな感じで動かします。例としてEthane。デフォルトはCPUに優しいSTO3Gの一点計算です。
import gamess g = gamess.Gamess() obc = ob.OBConversion() obc.SetInFormat("mol") mol = ob.OBMol() next = obc.ReadFile(mol, "examples/ethane.mol") print g.gamess_input(mol) try: newmol = g.run(mol) except GamessError, gerr: print gerr.value print newmol.GetEnergy() print [(obatom.GetIdx(), obatom.GetType(), obatom.GetPartialCharge()) for \ obatom in ob.OBMolAtomIter(newmol)]
結果はこれ。
$contrl runtyp=energy scftyp=rhf $end $basis gbasis=sto ngauss=3 $end $SYSTEM MWORDS=30 $END $DATA 6324 C1 C 6.0 -0.7560000000 0.0000000000 0.0000000000 C 6.0 0.7560000000 0.0000000000 0.0000000000 H 1.0 -1.1404000000 0.6586000000 0.7845000000 H 1.0 -1.1404000000 0.3501000000 -0.9626000000 H 1.0 -1.1405000000 -1.0087000000 0.1781000000 H 1.0 1.1404000000 -0.3501000000 0.9626000000 H 1.0 1.1405000000 1.0087000000 -0.1781000000 H 1.0 1.1404000000 -0.6586000000 -0.7845000000 $END -78.30530748 [(1, 'C3', -0.16967199999999999), (2, 'C3', -0.16967199999999999), \ (3, 'HC', 0.056557999999999997), (4, 'HC', 0.056559999999999999), \ (5, 'HC', 0.056554), (6, 'HC', 0.056559999999999999), \ (7, 'HC', 0.056554), (8, 'HC', 0.056557999999999997)]
ラジカルの計算がしたいのでROHFかUHFの設定ができるようにしておいたがスピン多重度の指定が出来ないのでとっととやる。
あと、テスト書かなあかんなぁと思いながらエキスパートPythonを読んでます。二周目か三周目かわからんけど、何回読んでもこの本は楽しい。
18062011 chemoinformatics Python
clone的なメソッド用意されてないのね。
この前は、こんな感じでコピーするようにしたけど、
def clone(mol): new_mol = ob.OBMol() for atom in ob.OBMolAtomIter(mol): new_mol.AddAtom(atom) for bond in ob.OBMolBondIter(mol): new_mol.AddBond(bond) return new_mol
適当なフォーマットに書きだして再読込でもいいそうなのでそうした。
def clone(mol): obc = ob.OBConversion() obc.SetInAndOutFormats("mol", "mol") molstring = obc.WriteString(mol) new_mol = ob.OBMol() obc.ReadString(new_mol,molstring) return new_mol
Flaskにはjsonifyって関数があるから、いま作ってるサービスをRESTにしようと思ったんだけど細かいところでうまい実装が思いつかなかったり、そもそもきちんと理解してないことも発覚してRESTful Webサービスを読み直している。
とりあえずJSON用のRESTを実装して、クライアントとしてのwebはそっちをアクセスするようにしたほうがいいのかなぁと。
ただ、そうするとURIがぶつかるからJSON用のAPIのほうは/v1/をpathの先頭につけたけど。
あと、FlaskのjsonifyってSqlalchemyの結果をそのまま渡すと駄目で、dictionaryを組み立てて渡さないとあかんのね。これがちょっとめんどくさい
7章のブックマークサービスをRoRで実装する章をFlaskで再実装するという修行をしないとあかんのかなぁ、、、
こんなふうにif-elseで振り分けるのと
@app.route('/some_path', methods=['GET,POST']) def some_method(): if request.method == 'POST': // POST用の処理 else: // GET用の処理
こんな風に明示的に分けるの
@app.route('/some_path', methods=['GET']) def get_some_path(): // GET用の処理 @app.route('/some_path', methods=['POST']) def post_some_path(): // POST用の処理
前者はインデントが深くなっちゃうので後者のほうが読みやすいと思うんだけど。
MLのアーカイブにあった
13062011 chemoinformatics Python
代謝部位を見積もるためには水素原子のBDE(結合解離エネルギー)を計算してラジカルになりやすい位置を探せばよいが、化合物毎に全ての水素の引き抜きエネルギーを求めるので計算用のインプットを作成するのに手間がかかる。
それをopenbabelでやってみたという話。これにGamessラッパーを組み合わせれば計算できそうな感じ。ケミストが使えるようにwebのサービスにすんにはまだまだ色々やらないといけないけど。
CYPの代謝予測っていうのは、分子認識+触媒メカニズムをきちんと理解しないといけない。後者は上で書いたような話で、前者はドッキングシミュレーションとかそういう話。触媒反応を理解しないでドッキングシミュレーションにばっか頼ると擬陽性が多くなるし、反応論に傾倒すると偽陰性が増えるので組み合わせが重要ってことで。
逆にそこら辺をきちんと抑えていると、単に代謝サイトをブロックするだけじゃなくて全然違うところから分子認識に干渉するために小さい置換基を導入して代謝ブロックしたりとか、おーメディシナルケミストやー!的な感動があったりする(他社だけどな)。
11062011 Python
シェルだったら
find . -name '*~' -print | xargs rm
に適当なaliasきっとけばいいんだけど、pythonで。
import os import re end_with_childe = re.compile('.*~$') for root, dir, files in os.walk('.'): for file in files: if end_with_childe.match(file): os.remove(os.path.join(root, file))
pep8をGrowlで通知させるようにして数日経つが、Emacsを左右に分割するようになった(今までは上下に分割)
pep8の一行79文字以内におさめるという規約のおかげで左右に分割すると読みやすい。
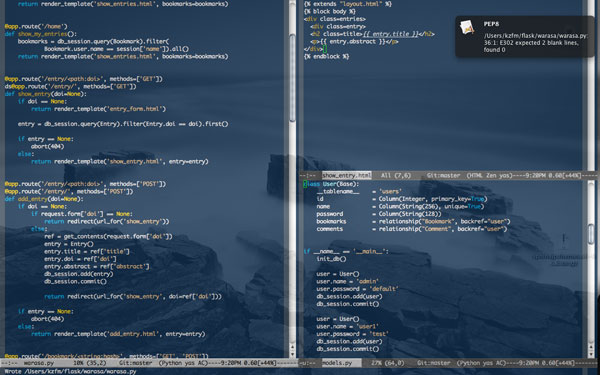
ちょっと生産性が上がった感がある。
07062011 Python
pep8ってコマンドが便利すぎてちょくちょく叩くんだけど、それでも叩くの面倒になったりすることはある。正直、こういうチェッカーは空気を読んで走って欲しいですね。
ユニットテストに組み込んでるのを見つけて、おーいいじゃん素敵じゃんとか思ってたんだけどもうちょい身近に感じたいなぁと。
で、Growlの出番。セーブするたびにpep8チェックが走って結果が通知されるようにした。
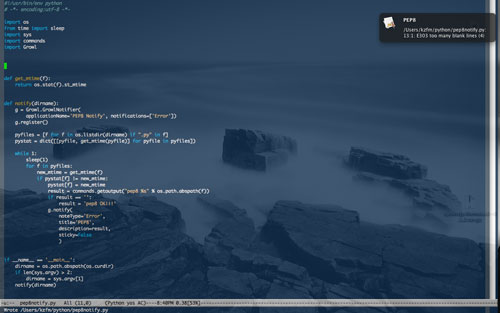
でもこの通知はすぐに消える(はやすぎ)。どこがおかしいのかよく見ようとすると消えてしまってちょっと困る。逆にstickyをTrueにすると消えずに残っているのでそれはそれでうざい。
神の御加護ってのはそんなもんなのかなぁと思った。あと、アイコンをPythonの蛇ロゴにしたほうがいいかなぁと。
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 | #!/usr/bin/env python # -*- encoding:utf-8 -*- import os from time import sleep import sys import commands import Growl def get_mtime(f): return os.stat(f).st_mtime def notify(dirname): g = Growl.GrowlNotifier( applicationName='PEP8 Notify', notifications=['Error']) g.register() pyfiles = [f for f in os.listdir(dirname) if ".py" in f] pystat = dict([[pyfile, get_mtime(pyfile)] for pyfile in pyfiles]) while 1: sleep(1) for f in pyfiles: new_mtime = get_mtime(f) if pystat[f] != new_mtime: pystat[f] = new_mtime result = commands.getoutput("pep8 %s" % os.path.abspath(f)) if result == '': result = 'pep8 OK!!!' g.notify( noteType='Error', title='PEP8', description=result, sticky=False ) if __name__ == '__main__': dirname = os.path.abspath(os.curdir) if len(sys.argv) > 2: dirname = sys.argv[1] notify(dirname) |
で、pep8走らせると長いStringが長すぎ!ってよく怒られるんだけど、長いURLとかしょうがないじゃん、無理して分割しても読みにくくなるじゃんと思うんだけどどうしたらよいんだろうか?