15042010 PRML
4.2にちょっと感動。
ロジスティック回帰で出てくるオッズ比はまぁそんなもんなのかなとかいって天下り的に覚えてたんだけど。
クラス分類の事後確率から考えていくほうが僕にとっては自然だ。
2クラスの分類問題(活性ありなしとか)を考えると事後確率は以下の式で表される。
分子で割ると
但し、αは以下の式で表される。
これはロジスティックシグモイド関数となる。
15042010 PRML
14042010 PRML
パターン認識と機械学習を読み直していて今4章。
図4.7のパーセプトロンのイメージが大変わかりやすいことに気づいて感激したのであった。
いまは、Pinheiroを読むのに苦労している。
 Mixed-Effects Models in S and S-PLUS (Statistics and Computing)
Mixed-Effects Models in S and S-PLUS (Statistics and Computing)これをきちんと理解しないと、PK-PDが進まないんだよな。
 Pharmacokinetic-Pharmacodynamic Modeling And Simulation
Pharmacokinetic-Pharmacodynamic Modeling And Simulation連休は旅行いったりとか子供と遊んでたんだけどそれ以外はJAGSいじってた。
5章をWikiに書いていくことにした。
分散分析のあたりを出来るようにしておくとよいと思った。
あとこの本だけだと難しすぎるのでPRMLの下巻が必須(多分)。
水産資源学におけるベイズ統計の応用ワークショップのスライドの22,23枚目
となっているのだが、23枚目のRのコードは
n2 <-sample(length(x1), 1000, replace=T, prob=dlnorm(x1,1.1,0.6))
とwを求めずにいきなりサンプリングしているので?となったが、一様分布だから重みを計算する必要ないのね。
わざわざ書くならこうか。
x1 <-runif(100000,0,60)
w <- dlnorm(x1,1.1,0.6) / (dunif(x1,0,60))
n2 <-sample(length(x1), 1000, replace=T, prob=w)
x2 <-x1[n2]
plot(density(x2))
15082009 Python matplotlib PRML
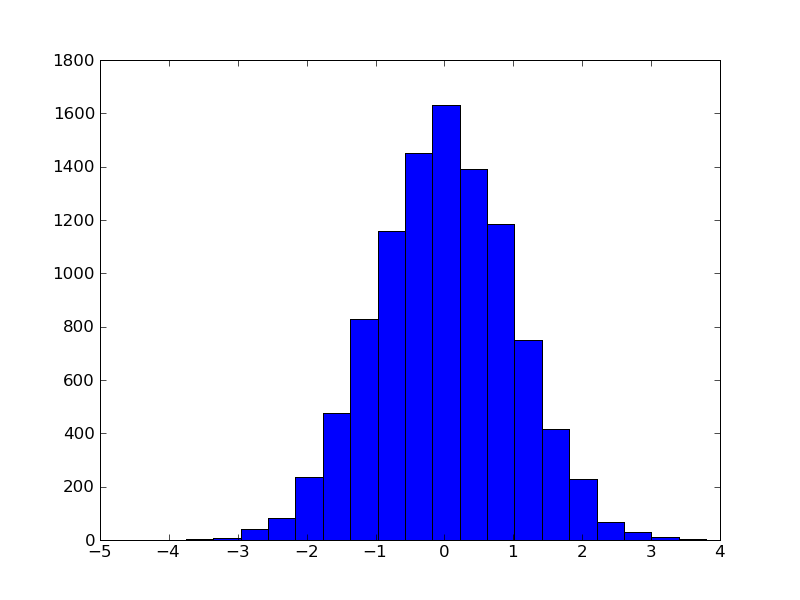
PRML11章。一様乱数から正規乱数を発生させる。
from math import sin,cos,pi,log,sqrt
from random import random
def box_muller():
z1 = random()
z2 = random()
return sqrt(-2*log(z2)) * sin(2*pi*z1)
if __name__ == "__main__":
from pylab import *
x = [box_muller() for i in range(10000)]
hist(x,20)
savefig("box_muller.png")
13082009 chemoinformatics R PRML
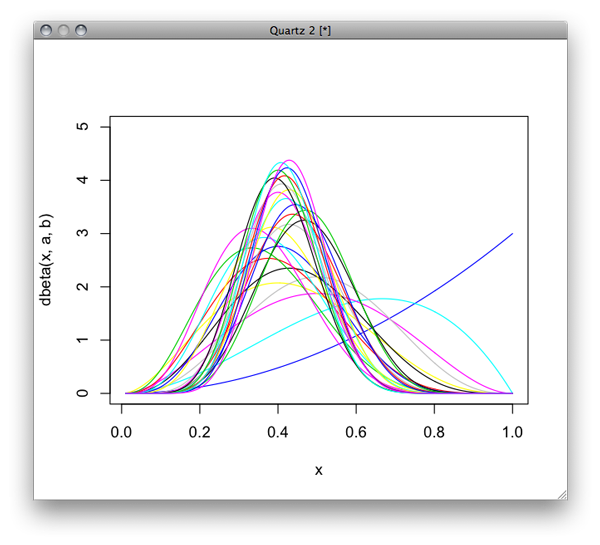
PRMLも再読してるが、手動かしながら読むほうがしっくりくる。 p.70のベータ分布の逐次学習ってのが面白そうなのでやってみた。
data <- rbinom(30,1,0.5)
plot_dbeta <- function(data){
a <- 0
b <- 0
x <- seq(0.01, 1.0, len = 500)
for (i in 1:length(data)) {
if(data[i] == 1) { a = a + 1 }
else { b = b + 1 }
if(i>3){
if(i==4){ plot(x, dbeta(x,a,b),type = "l",col=i,ylim=c(0,5)) }
else { plot(x, dbeta(x,a,b),type = "l",col=i,xlab="",ylab="",axes=F,ylim=c(0,5)) }
par(new=T)
}
}
}
二項分布で30個のリストを発生させて読みながらa,bのデータを更新していきつつその時のベータ分布の密度を求めつつプロットしていくという操作。
> data
[1] 1 0 1 1 0 0 0 1 0 0 0 1 0 0 1 1 1 0 1 0 0 0 1 0 0 1 0 1 0
[30] 1
データはこんな感じで、4番目のデータからプロットしていくとだんだん二項分布に近づいていく様がみてとれる。
15112008 chemoinformatics bioinformatics PRML
chemoinformaticsにも使えそうなので色々読んでみている。
ぼんやりとした入門のそのまた入門の入り口ぐらいには立ったかなって感じ。CRPで遺伝子発現のクラスタリングをやっている論文があったので読む。
ディリクレ分布に関してはこの本見た。
昨日気になったので、とりあえずどんな感じか確かめようと。
> library("fastICA") > library("mclust") > data("iris") > b <- fastICA(iris[1:4],2) > mc <- Mclust(b$S,G=3,modelNames="VVV") > plot(mc,b$S)
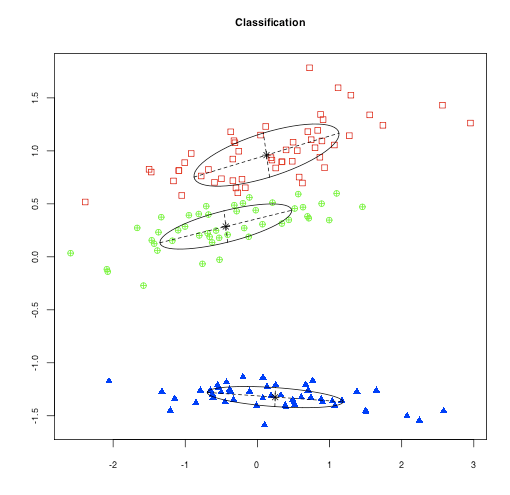
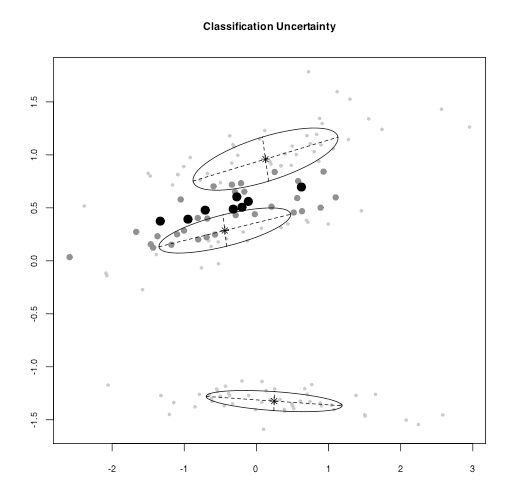
混合分布はクラスタリングの本にも載っているが、PRMLの下巻のほうがK-meansからEM,混合モデルの導入の流れがスムーズで分かりやすかったかな。