08112016 food
中華街も色々探索したいところ
07112016 food
コンセプトが見えない本だなと思ったらしずおか食セレクションを集めただけだった。
06112016 book
ザラッとした感じが心地よい
05112016 food
料理の四面体という本がとてもよかった。
料理をするということは、 火を利用し何らかの物質(液体または気体)を媒介させて熱を加える という単純明快なプロセスモデルで説明できるという主張であった。
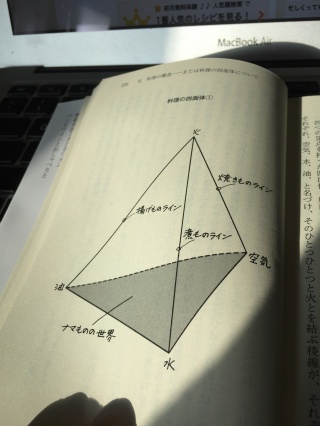
要するに、水、油、空気の三点を頂点とする三角形を底面として、火を頂点とする四面体のどこかに料理というモノは位置づけられるというのが筆者の言いたいことである。
料理を経験していくと材料のアレンジ(じゃがいもを里芋に変えてみる、醤油を煎り酒や魚醤に変更してみる)は上手くなっていくように思う。本書を読むと材料でなくプロセスを上手く変更するためのヒントを得やすくなるのではないかと思った。
料理を経験していくと材料のアレンジ(じゃがいもを里芋に変えてみる、醤油を煎り酒や魚醤に変更してみる)は上手くなっていくように思う。本書を読むと材料でなくプロセスを上手く変更するためのヒントを得やすくなるのではないかと思った。
具体的な例として思いつくのは次の本に載っていたレシピだ。かんたんで美味しいのでたまに作る
これだけなのだが、これってアヒージョの変形じゃないかと気がついたのが、アヒージョを作るようになってからだいぶ経ってからなのだw 気がついたときにはちょっと感動した。
といったようなことを考えると、底面の三角形は実はもっと曖昧なものなのではなかろうかとも思えてくる。結局水の沸点が100度なので
という温度帯で液体のくくりにしてもいいのではないかと思う。水で揚げ物は出来ないからね。
と書いておいて、ヘルシオ(ウォーターオーブン)あるじゃん。アレ使えば油なしで唐揚げできるじゃんと。
03112016 pokemongo
ハロウィン期間中に高個体値ヒトカゲを無事にリザードンまで進化できた。
CP18のリザード連れて歩いてたら、2kmたまごから最高コメントのヒトカゲ出てきて今飴が114。駄目なリザード進化して図鑑を埋めるか、あと11個飴を集めて最高のリザードン作るか悩む。尚リザードンに思い入れはあまりない…
— kzfm (@fmkz___) 2016年11月1日

あとはプテラ、サワムラーの二種だけど「千本浜」や「三保の松原」に篭もるのもなんか面倒くさいので10kたまごを割るために歩き回ることになりそうだ。
goプラスほしいなぁ
03112016 food
餃子
どうでもいいことなので書く必要があるのかどうか悩んだけど、SNSでつながっていなくてblogをチェックしているヒトが何人もいるので一応こっちにも書いておきます。
今日から久しぶりの電車通勤をしているので沿線沿いの方々飲みに誘ってください。そしてmacbookを抱えて通勤するのでアルコール注入しながらのハックをしましょう。
01112016 book
一話はTVで見たことがあった。科学ミステリー
ホンワカ感漂う
勢いがあるが、あまり好みじゃないので表題作だけ読んだ。悪くはないと思う
29102016 work
ユーザー会お疲れ様でした。ヒトのつながりが複雑に絡まり合っていることを再確認した。演題はそれなりに面白いものばかりだったけど、ぶっ飛んでいる感じのするものは見当たらなかったが、新陳代謝が活発なので今後に期待。
木曜日は最近お気に入りのエリックサウスに行ったら、ビリヤニ売り切れてて、ランチミールスをいただいた。ラッサム、サンバル、パパドと主要キーワードは覚えた。

残念すぎたので次の日の金曜日は開店凸してビリヤニをw ビリヤニ美味しいですね。尚、カレーに混入している黒い小さな粒は何の種だか気になったけどわからなかった。自分のカレーでは使ったことないからスゴイきになる。

そして、土曜日はどうしても行きたかった形成立石の宇ち多゛に行ってきた。サイダーをチェイサーにしても梅割り二杯しかいけなかった。活気というか雰囲気が好きな感じで楽しめた。
煮込み、お新香、タン生。
煮込みも美味しいけどタン生美味すぎた…

シロタレ、アブラタレ。シロタレ好み、アブラはあぶらぎった旨さw

ガツ塩

レバタレ

注文するのに呪文を覚える必要があるんだけど、他の人の注文をなぞればいいので割りとかんたんだった。カシラとハツが売り切れていたので次回は開店凸してゲットしたい。
近所の栄寿司の行列もやばかった