06052019 life
近所の公園でラーメンフェスタを開催していたので、ちょっと覗いてきた。
静岡のラーメン屋さん。行列が少ないところにしてみた。


長野のラーメン屋さんで醤油ラーメン。子供が食べてた。


仙台のラーメン屋さん。牛タン塩ラーメン


ついでに神社でもライブがあったので、寄ってきた。


06052019 life
近所の公園でラーメンフェスタを開催していたので、ちょっと覗いてきた。
静岡のラーメン屋さん。行列が少ないところにしてみた。


長野のラーメン屋さんで醤油ラーメン。子供が食べてた。


仙台のラーメン屋さん。牛タン塩ラーメン


ついでに神社でもライブがあったので、寄ってきた。


01052019 English
サマー先生と英会話 !というyoutubeチャンネルをたまに見ているので、アフィリエイト料が入った流れで買ってみた。
part1はなかなかおもしろかったけどpart2のセンテンス集は基本的過ぎたので、itunesに突っ込んで流し聴きするくらいでよいかなと思った。
もともと娘に渡すつもりで買ったので特に問題はなかった。
29042019 bioinformatics
理解した
28042019 chemoinformatics bioinformatics
医薬品研究開発における Modeling and Simulation(M & S)手法の紹介という大変に面白い総説を教えてもらって読んでいる。
これは、事前にTrkAがpainのターゲットであることを知っているからこういう結論に出来たのか、そういう事前知識なしに自然に導かれたのか興味あるところではある。モデルからターゲット候補が出てくるなんて素敵。
QSP モデルによって,今までのモデル解析ではできなかった予測を行った事例を以下に 2 例紹介する.1 つ目は,MID3 白書に取り上げられている神経成長因子(NGF)パスウェイに対する QSP 解析 により,疼痛治療の新規の創薬ターゲットを検討した事例(Example 3)である.この事例では,すで に報告されていた NGF パスウェイを ODE で記述される数理モデル(Model 1)に変換し,さらにその パスウェイモデルを関連因子の神経細胞内外での挙動を反映させたモデルに連結して生理学的に拡張し たモデル(Model 2)を作成して NGF パスウェイの下流での創薬ターゲット候補の探索を行っている. 効果測定の指標として NGF 刺激の結果として観察され,疼痛発現につながると考えられる Diphosphorylated extracellular signal-regulated kinase(dppERK)の蓄積量を用い,モデルの妥当性検討は すでに報告されている NGF 阻害抗体で得られている結果と比較することにより行った.こうして構築 されたモデル内に含まれる因子を,in silico で仮想的に様々な阻害強度で阻害する感度分析により, Tropomyosin receptor kinase A(TrkA)が薬物治療のターゲット候補となる可能性が予測された.
尚、MoAと書くと現状はBabymetalがおすすめされるようです。
 「LEGEND - S - BAPTISM XX - 」 (LIVE AT HIROSHIMA GREEN ARENA) [Blu-ray]
「LEGEND - S - BAPTISM XX - 」 (LIVE AT HIROSHIMA GREEN ARENA) [Blu-ray]PK-SimってOSSになったの?
27042019 chemoinformatics bioinformatics
最近、バイオインフォマティクスのチームに兼務になりました。このバイオインフォマティクスチームに求められてることが、いわゆる普通のターゲットファインディング的なバイオインフォだけじゃなくてPKPD(今だとQSPって言うの?)でのインフォマティクス側からの貢献を求められたりするので10連休は色々勉強するよいチャンスだったりします。しかし、PKモデリングは基本的なところはわかるけど、PDは全然だから着いていくのつらいっす。
それから、組織内でモダリティのブームが起きているみたいなので、そっちのほうも色々キャッチアップしなきゃいけない。
で、自分の解釈でいうところのモダリティというのは、「従来のkey&lock型の低分子創薬を超えて、MOAをうまく利用して生体に干渉するような創薬をやりましょう」ってことだと思うんですよね。つまりライフハック。
そういう意味ではこれからの分子設計は低分子だけじゃなく、中分子や核酸やdegradationでも貢献する必要があるだろうし、創薬ターゲットのMOAに応じて柔軟に対応出来るような組織になっているべきでしょうね。と考えるとMOAを理解できることが必須になるんじゃないですかね。
というわけで、分子生物学も少し復習しないとなぁという気分になったのでそれっぽい本を読んでいました。遺伝子ドライブ面白いですね。
コラムにCRISPRdirectが紹介されていて、「あー作者の人知っとるわ」ってなった。
13042019 chemoinformatics
創薬プロジェクト用のIssue Tracking System(ITS)を探しているのだけど良いものがみつからない。
で、ないのなら作ってしまえということでとりあえずE-R図でも書こうかとnode.jsインストールしてwwwsqldesignerでやってみた
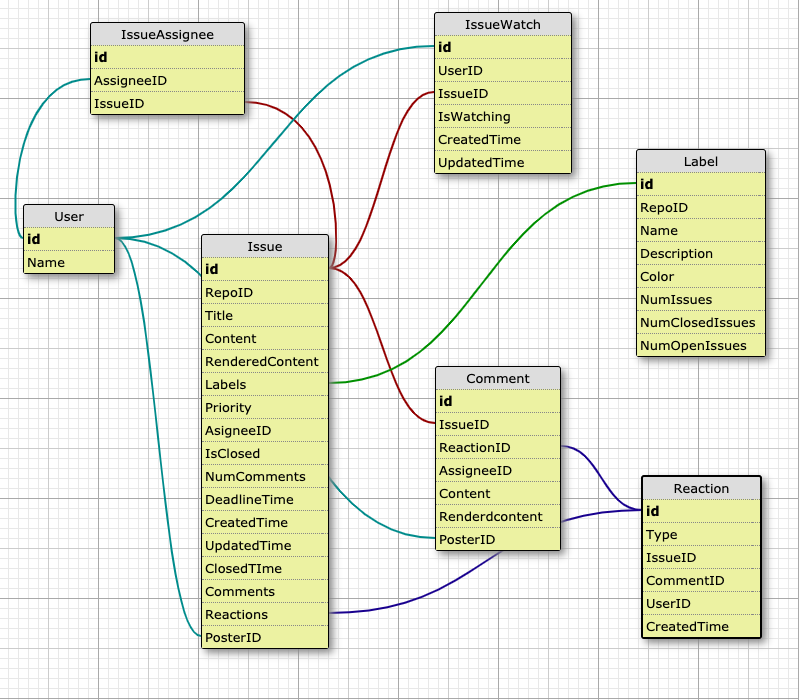
テーブルスキーマ出来たんであとはFlaskでサクサクつくればよろしい。
先週行ってきた

桜



よい


帰りは三島二日町まで降りてきて隣の田町駅の向かいにあるやんぐでラーメンを食べました。

11042019 三島
チームから1人離脱するので、やごみで送別会的な飲みをしてきました。所属が離れるけど兼務でつながってるし同じフロアにいるので離れた感はゼロだけどねw

湯豆腐とマグロ


アジと定番の磯辺揚げ


タコ唐揚げがつまむのに良かった

今年もみんなで楽しく仕事をして良い成果を出せるといいなーと思います。