SICPの4章とか読み始めたらDSLが気になってきてJesse Vincent - Abusing Domain Specific Languages for Fun and Profitをちゃんと見ようと。
で、気付いたんだがgoogle video playerってどうなっちゃったんだろうか?
まぁいいかってことで、mp4で落とそうとすんだけどなぜか10Mぐらいでこけてしまう。
SICPの4章とか読み始めたらDSLが気になってきてJesse Vincent - Abusing Domain Specific Languages for Fun and Profitをちゃんと見ようと。
で、気付いたんだがgoogle video playerってどうなっちゃったんだろうか?
まぁいいかってことで、mp4で落とそうとすんだけどなぜか10Mぐらいでこけてしまう。
位置を指定するとその位置のパスカル三角形の値を返す。再帰で。
sub pascal{
my ($n,$r) = @_;
$n<2?1:$r==0?1:$n==$r?1:pascal($n-1,$r-1)+pascal($n-1,$r);
}
print pascal(0,0);
print "\n";
print pascal(1,0);
print pascal(1,1);
print "\n";
print pascal(2,0);
...
print pascal(4,3);
print pascal(4,4);
print "\n";
三角形
$ perl pascal.pl
1
11
121
1331
14641
この関数を利用して、最小完全ハッシュ関数を。
sub cn {
my @table = @_;
my ($hash,$n,$r) = (0,5,2);
for (my $i=0;$n>$r && $r>0;$i++){
$n--;
$hash += pascal($n,$r--) if ($table[$i]);
}
return $hash;
}
が、「組合せ型の最小完全ハッシュ関数」の逆関数に持っていけなかった。再帰でパスカル三角形を右斜め下から左斜め方向に足していく組み合わせをつくっていって問い合わせの数値になったところのリストを返せばよいと思うんだけど、うまい書き方がみつからなかった。
21082007 chemoinformatics perl
職場でいつものようにGUIをチマチマいじってたら、隣で塩を取り除くコマンドがないっちゅよと困っていた。openbabelにあるんじゃねーの?どれどれと探してみたけどなかった。さらに、いやあるだろふつーはとかいってググってもcgiみたいなもんしかみつからなかった。
smilesだったら.でsplitしてゴニョればOKなんだけど、座標情報落っこちるから、perlmolとかopenbabelで実装して任意のフォーマットにできるようなコマンドが欲しい。
それを仮にwashmolとすれば
washmol -i input.sdf [-o out.smi]
みたいに。デフォルトではインプットと同じフォーマットで返すような、調子いいコマンドが欲しくなった。
僕は塩を取り除くのはwashと言ってるんだけど、desaltとか色々と呼び方があんのね。
20082007 perl
動かないのでこっちに書いておく。
どう書く?のお題でPageRankの計算を解いてたんだけどうまく動かない。
use Math::MatrixReal;
my $data = {
1 => [2, 3, 4, 5, 7],
2 => [1],
3 => [1, 2], 4 => [2, 3, 5],
5 => [1, 3, 4, 6],
6 => [1, 5],
7 => [5],
};
sub pagerank {
my $data = shift;
my $rows = scalar(keys %$data);
my $mat = Math::MatrixReal->new($rows,$rows);;
while(my ($i, $link) = each %{$data}){
for my $j (@{$link}){
$mat->assign($j,$i,1/scalar(@{$link}));
}
}
#print $mat;
$det = $mat->det();
print "det: ",$det,"\n";
}
pagerank($data);
実行結果
$ perl pr.pl
det: -0
detが-0ってことは、複素数の計算ができてないとかそういうことかなぁ。全然わからんので書きかけで放り投げた。
今週は朝も夜も週末も集中して読めたため、2章を読み終えた。最後の2.5はなんかいい加減に読んだので後々読み返すことになりそうな気もするが。
p.109のメッセージパッシングとOOPって関連するよな。と思って調べてみたけどいまいちうまい表現が見つからなかった。
あとperlでOOPやるときのblessとmy $self = shift;みたいに第一引数が自分自身なのとperlのオブジェクトがblessされたハッシュリファレンスなこととか、もうすこしでちゃんと理解できそうな感じなんだけど、Perl におけるオブジェクト指向を読んでちょっと前進。
おもしろいですな、SICP
17082007 perl
最近朝起きてSICPを読んでる。で、SICPの2章も中盤を越えて、accumulateを使って行列の演算を定義した。
ふむーこれはpythonでやってみたいなと思ってたら、ちょうどタイミングよくpythonでやってる人がいた。
ので、perlで。
sub accumulate{
my ($op, $init, $list) = @_;
@$list == () ? return $init :
return $op->(shift(@$list),accumulate($op,$init,$list));
}
my $test_func = sub {return $_[0]+$_[1];};
my $test_list = [1,2,3,4,5];
print accumulate($test_func, 0, $test_list);
で、SICPみたいに、たたみこむだけじゃなくてリストを戻したい。
my $cons = sub {return [@{$_[1]},$_[0]];};
XXX accumulate($cons, [], [1,2,3,4,5]);
List::Utilでも同じことできるみたいですが。自分で書いてみたかったということで。
16082007 perl
12082007 chemoinformatics perl bioinformatics
さて、bioinformaticsとかchemoinformaticsとか言われてるようなアレは、化学とか生物学に対して情報学的観点からアプローチしたりするわけです。こういう点から見ると、薬ってもんは蛋白質にうまいことはまる鍵みたいなもんで、蛋白質の穴にいい感じではまるような化合物を(コンピュータを駆使して)設計していくのが(コンピュテーショナルな)ドラッグデザイン(CADD)という分野だヨ。
で、蛋白質というものは複数のアミノ酸から構成され(100から数百)そしてアミノ酸は20種類程度存在し、それぞれ数十の原子から構成されているわけだ。要するに一対多の階層構造をとる。
protein -> amino-acid -> atom
みたいな。
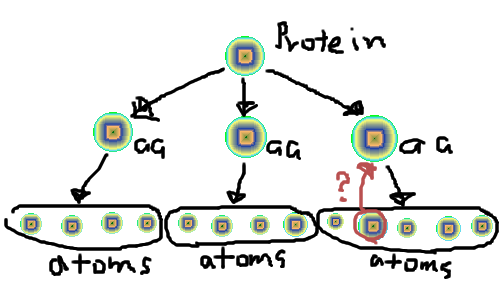
でそれぞれprotein aminoacid(aa) atomみたいなクラスを用意すれば
$atom1 = Atom->new({name => 'C1', type => 'C'});
...
$aa1 = AminoAcid->new({name => 'GLY', atoms => [$atom1, $atom2, ]})
...
$protein = Protein->new({name=> 'ProteinA', aminoacids => [$aa1,$aa2,$aa3...]});
みたいにそれぞれ配列に突っ込めば蛋白質を表現できて、あるatomオブジェクトを与えられた場合にそれがどのアミノ酸に属しているのか調べるのに
for my $aminoacid ($protein->aminoacids){
for my $atom ($aminoacid->atoms){
return $aminoacid if $qatom == $atom;
}
}
みたいにdepthfirstで探索していけばいいんだろうけど、ちょっと探索効率が悪いので、atomオブジェクトに $atom->{parent} = $parent_aminoacidみたいに親のアミノ酸オブジェクト返すような属性追加したんだけど、これだと構造が複雑になってなんか気持ち悪い。
他にうまいやり方ってあんのかなと思ったお盆の夏2007。
10082007 chemoinformatics perl
OSRAがええよとか言ってたわけだが、そもそも画像のメタデータに構造情報埋め込めばいいやん的発想なのがこのエントリ。
use Image::ExifTool qw(:Public);
use XXX;
$info = ImageInfo('rosiglitazone.png');
XXX $info;
で実行
$ wget http://depth-first.com/demo/20070801/rosiglitazone.png
$ perl etest.pl rosiglitazone.png
---
BitDepth: 8
ColorType: RGB with Alpha
Compression: Deflate/Inflate
Directory: .
ExifToolVersion: 6.90
FileModifyDate: 2007:08:01 21:18:16
FileName: rosiglitazone.png
FileSize: 8 kB
FileType: PNG
Filter: Adaptive
ImageHeight: 109
ImageSize: 327x109
ImageWidth: 327
Interlace: Noninterlaced
MIMEType: image/png
PixelUnits: Unknown
PixelsPerUnitX: 1
PixelsPerUnitY: 1
molfile: |-
name
params
comments
25 27 0 0 0 0 0 0 0 0 0 V2000
1.6910 -6.1636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5571 -6.6636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4231 -6.1636 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
3.4231 -5.1636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5571 -4.6636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6910 -5.1636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2891 -4.6636 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
...
23 19 1 0 0 0 0
22 24 2 0 0 0 0
20 25 2 0 0 0 0
M END
...
at etest.pl line 6
論文のpdfとかもこういう感じで画像を埋め込んでくれればちょっとはデータが取り出しやすくていいかも。