演習の本をRでこなしていくことにした。
設問1
β層の消失速度をもとめて、それを用いてα層の消失速度を決定する。
t <- c(1,2,3,6,13,23,30,39,49)
c <- c(68.2, 51.1, 43.2, 34.0, 18.3, 11.5, 10.7, 10.6, 6.78)
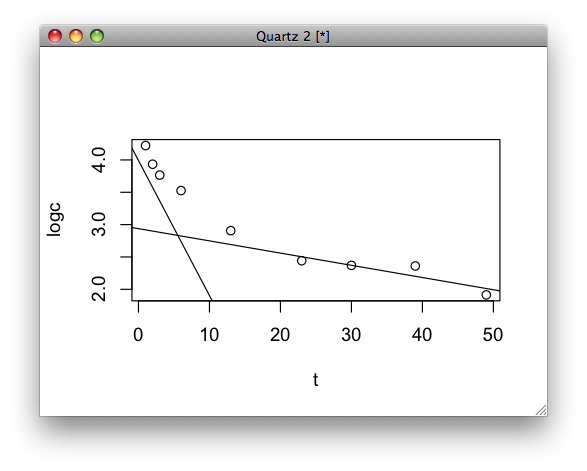
23時間からの濃度からβ層の消失速度を求める
logc <- log(c)
result <- lm(logc[6:9] ~ t[6:9])
coef(result)
(Intercept) t[6:9]
2.93770720 -0.01888941
β層の消失速度が決まったので、次は23時間までの濃度からβ層の寄与を引いた濃度からα層の消失速度を決定する
beta <- function(t){exp(2.937707-0.01889*t)}
alogc <- c[1:5] - beta(t)[1:5]
aresult <- lm(alogc ~ t[1:5])
これでα層β層それぞれの消失速度がもとまる
result
Call:
lm(formula = logc[6:9] ~ t[6:9])
Coefficients:
(Intercept) t[6:9]
2.93771 -0.01889
aresult
Call:
lm(formula = alogc ~ t[1:5])
Coefficients:
(Intercept) t[1:5]
3.9916 -0.2088
求めるモデルは
exp(3.9916-0.2088*t)+exp(2.937707-0.01889*t)
設問2
ラフに求めたA,B,alpha,betaを初期値として最小自乗法でフィッティングを行う。1exp,2expでやってみて、どっちのあてはまりがよいか考える
t <- c(1,2,3,6,13,23,30,39,49)
conc <- c(68.2, 51.1, 43.2, 34.0, 18.3, 11.5, 10.7, 10.6, 6.78)
dat <- data.frame(conc,t)
result <- nls(conc ~ a*exp(-alpha*t),start=list(a=50,alpha=0.045))
summary(result)
Formula: conc ~ a * exp(-alpha * t)
Parameters:
Estimate Std. Error t value Pr(>|t|)
a 63.26990 5.39879 11.719 7.45e-06 ***
alpha 0.08359 0.01777 4.706 0.00219 **
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Residual standard error: 6.608 on 7 degrees of freedom
Number of iterations to convergence: 14
Achieved convergence tolerance: 7.74e-06
f <- function(t){63.2699*exp(-0.0836*t)}
plot(f,0,50,xlim=c(0,50),ylim=c(0,70),xlab="Time",ylab="Conc")
par(new=T)
plot(t,conc,xlim=c(0,50),ylim=c(0,70),axes=F,xlab="",ylab="")
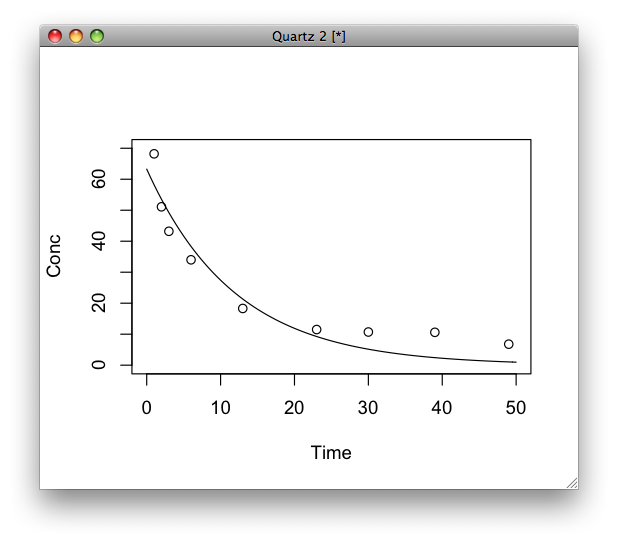
一層の消失モデルだと特に遅い時間の当てはめが悪い。ちなみにRだとAIC関数使えば適合度がでる。
t <- c(1,2,3,6,13,23,30,39,49)
conc <- c(68.2, 51.1, 43.2, 34.0, 18.3, 11.5, 10.7, 10.6, 6.78)
dat <- data.frame(conc,t)
result <- nls(conc ~ a*exp(-alpha*t)+b*exp(-beta*t),start=list(a=50,alpha=0.277,b=20,beta=0.0198))
summary(result)
f <- function (t){55.75*exp(-0.3769*t)+29.157*exp(-0.03189*t)}
plot(f,0,50,xlim=c(0,50),ylim=c(0,70),xlab="Time",ylab="Conc")
par(new=T)
plot(t,conc,xlim=c(0,50),ylim=c(0,70),axes=F,xlab="",ylab="")
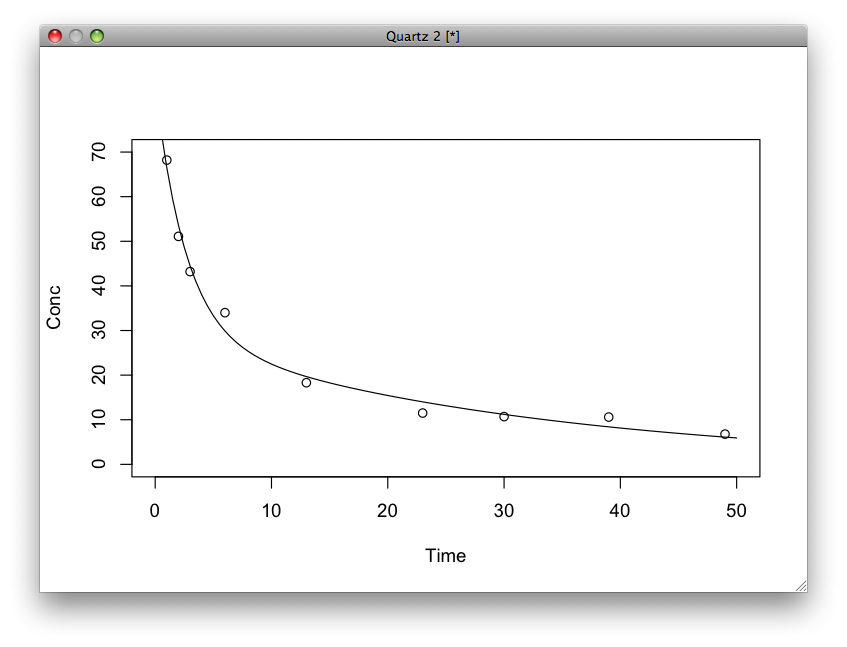
プロットするときにpar関数で上書きするのと、xlim,ylimを明示して軸をきちんとそろえつつ、目盛やタイトルを上書きして重ねないように気をつける。

 ファーマコキネティクス―演習による理解
ファーマコキネティクス―演習による理解
 Computer Applications in Pharmaceutical Research and Development (Wiley Series in Drug Discovery and Development)
Computer Applications in Pharmaceutical Research and Development (Wiley Series in Drug Discovery and Development) Analysis of Integrated and Cointegrated Time Series with R (Use R!)
Analysis of Integrated and Cointegrated Time Series with R (Use R!) Data Manipulation with R (Use R!)
Data Manipulation with R (Use R!) Bayesian Computation with R (Use R)
Bayesian Computation with R (Use R) Applied Spatial Data Analysis with R (Use R!)
Applied Spatial Data Analysis with R (Use R!) Analysis of Integrated And Cointegrated Time Series With R (Springer Series in Statistics)
Analysis of Integrated And Cointegrated Time Series With R (Springer Series in Statistics) Applied Statistical Genetics with R: For Population-based Association Studies (Use R!)
Applied Statistical Genetics with R: For Population-based Association Studies (Use R!) Analysis of Phylogenetics And Evolution With R (Use R)
Analysis of Phylogenetics And Evolution With R (Use R) Nonlinear Regression with R (Use R!)
Nonlinear Regression with R (Use R!) Wavelet Methods in Statistics with R (Use R!)
Wavelet Methods in Statistics with R (Use R!) Rで学ぶクラスタ解析
Rで学ぶクラスタ解析 集合知プログラミング
集合知プログラミング
 パターン認識と機械学習 下 - ベイズ理論による統計的予測
パターン認識と機械学習 下 - ベイズ理論による統計的予測