序盤から全然ダメだった。
27072014 bioinformatics Python
Bioinformatics版のProject Euler
RosalindというProject Eulerっぽいサイトがあったので少し遊んでみた。
楽しくbioinformaticsの問題を解きながらアルゴリズムを覚えていく感じなので、BioPythonを覚えるのにちょうどいいかなと思った。
GC Contentを計算するメソッドくらいあるだろうとは思うんだけど知らないので素朴に計算。リスト内包表記でmaxで取り出すのがいいかなと思ったけど、面倒くさいのでこれまたループで素朴に。
import sys from Bio import SeqIO def count_gc(seq): return float((seq_record.seq.count("G") + seq_record.seq.count("C"))) * 100 / len(seq_record.seq) if __name__ == '__main__': fasta = sys.argv[1] highest_id = "" highest_content = 0 for seq_record in SeqIO.parse(fasta, "fasta"): c = count_gc(seq_record) if c > highest_content: highest_id = seq_record.id highest_content = c print highest_id print "{0:.6f}".format(highest_content)
僕はBioPerlでバイオインフォマティクス関連のプログラミングをしてたので、BioPythonで配列解析をしたことは殆ど無いんですよね。
 Bioinformatics Programming Using Python: Practical Programming for Biological Data (Animal Guide)
Bioinformatics Programming Using Python: Practical Programming for Biological Data (Animal Guide)Mitchell L Model
O'Reilly Media / ?円 ( 2009-12-08 )
中飛車穴熊
ここのところ中飛車に出会う確率が高い
11手目はなんとなく6六銀としてみた。
22手目で相手が5一金と差してきたので「ん?穴熊か…」と。
端歩を突いたらやっぱり受けなかったので穴熊だった。
中飛車で穴熊って初めてかもと思いつつ、じゃぁ自分の金は攻めに使おうってことで、4六からすりつぶしにいった。
スリスリするのはいいんだけど穴熊だと駒がドンドン補充されてなかなか穴が掘れなくて焦った。自分の片美濃はちょっと攻められたら簡単に潰れちゃったし。
結局相手の攻め駒が無くなったところで投了
ゴキゲンな中飛車
ゴキ中で負け続けた結果、先手でも後手でも相手がゴキ中だと相ゴキ中にするようにしている。
ゴキ中は結局捌き合いなのかなと思ったりもする(片美濃だし)
尚、振り飛車をLLに例えると
- 三間飛車: Perl
- 四間飛車: Python
- 中飛車: Ruby
- 向かい飛車: Haskell
な感じですね。参考になればとw
21072014 Snorkeling
井田に潜りに行ってきた
7/19から海水浴場がオープンしたので早速井田に潜りに行ってきた。
西浦のらららは怖過ぎて撮影できないw あそこに行くくらいならもうちょっと遠征して江梨とかのんびりしたところがあると思うけどなぜあんな混むところに行きたがるのだろうか…?
途中の大瀬崎。そこそこ入っている感じでした。


井田は気温29度海水温24度ということだったが、水温は24度なかったはず(冷たかったw)
ふぐとかカワハギとかベラとかまぁ色々いて癒やされたが、水温低いと体力の消耗が激しいですね。もう少し経てば水温があがって快適に潜れるのではないかと。
尚、耳抜きがうまく出来なくてちょっと課題を残した1日であった。

井田はまったり出来ていいですね。それからフィンの威力を思い知った。
mewを履いています☆
20072014 chemoinformatics work
某WSお疲れ様でした
とても勉強になって参加して良かったなと。また今後のキャリアを色々と考えないとなーという話を沢山聞いた。ホント、そろそろどうするかちゃんとしないとなぁと思った。
それから、初めてChemRubyの作者の方とお話出来て良かったです(よく考えるとあたりまえだけどbonohuさんの知り合いだったw)
前回の勉強会のアンケートも揃ったし、次回を三島バルにぶつけるなら、お盆前に反省会と次回のテーマ決めを兼ねてやごみか鈴木屋に集まらないといけませんね☆
p.s.
bgは統合TVのスライドをslideshareにアップロードしておいて下さい
17072014 drum'n'bass
三間飛車急戦
仕掛けが早くて美濃に囲えなかった。しょうがないのでカニカニ銀みたいに銀を出していこうかなと考えたがやっぱりやめた。居玉なんてbg7860みたいじゃないか…w
潰されなかったので良かったけど。
自分でも石田流を少し指してみたい。
後は将棋始めた時に読んでさっぱり分からなかった四間飛車を指しこなす本を再度読んだら面白くてこればかり読んでいる。
08072014 chemoinformatics Python
あらかじめクラスタ数を決めないでクラスタリングする方法(Affinity Propagation)
K-meansのように予めクラスタ数を指定すると、「そのクラスタ数は正しいの?」っていう疑問が浮かぶと思う。
「なんらかの統計値に基づいて適切なクラスタに分割して欲しい」そんな願いを叶えるのがAffinity Propagationというクラスタリングアルゴリズムである
exemplara(セントロイドとかクラスタ中心)になるべきパラメータ(responsibility)とクラスタメンバに属しやすさ(availability)を交互に更新していって収束させる手法なので、K-meansのような初期値依存性がないらしい。
クラスタ数は類似度行列の対角要素(自分との類似度)に依存する(デフォルトはmedian)のでここを変更するとクラスタ数も変わるんだけどね。
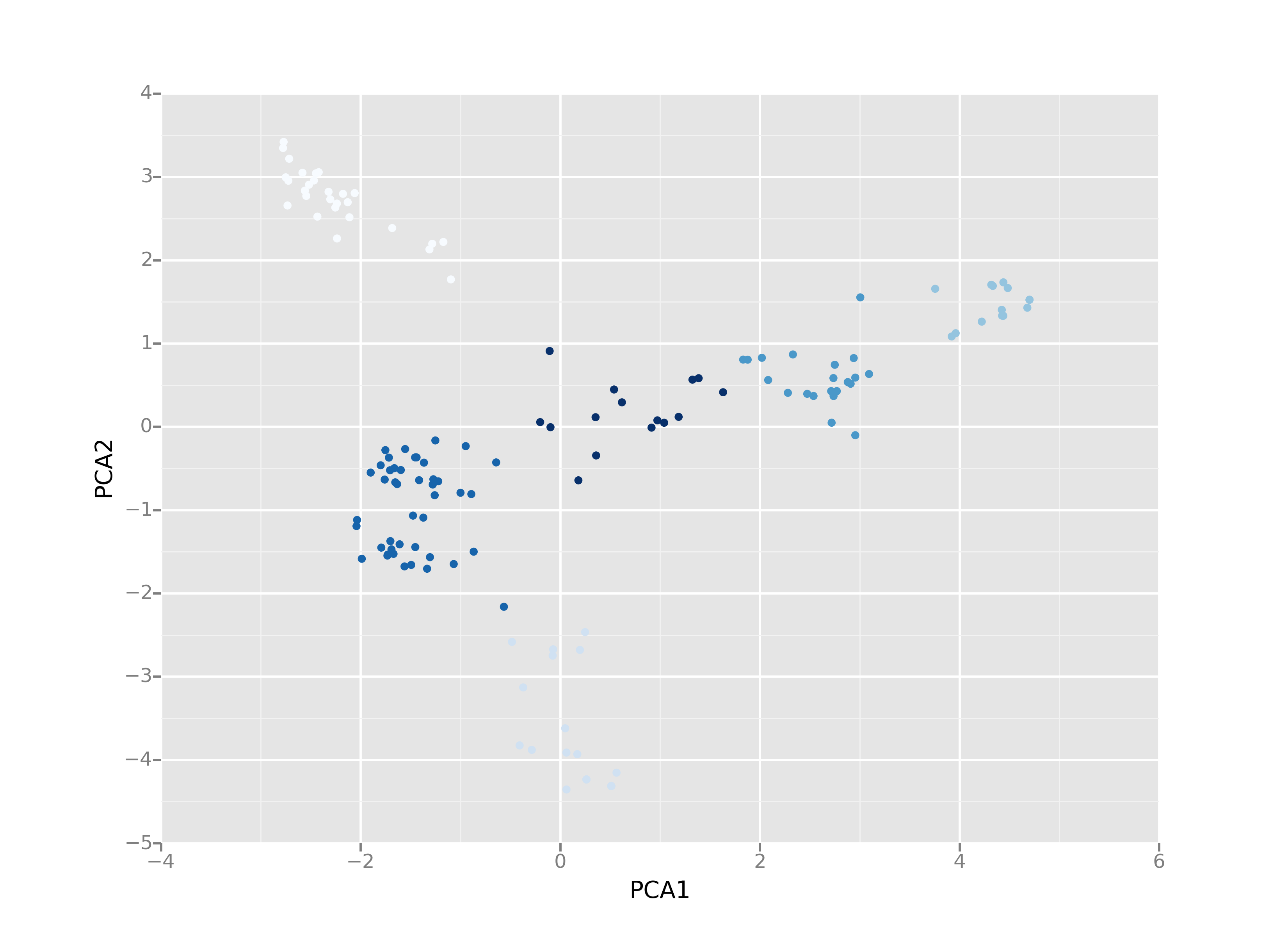
Scikit-learnではAffinity Propagationが実装されているのでsykのケミストリースペースを作ってクラスタリングしてみた。ちなみにスライドのPCAの説明は間違っていた(pca.fit(fps).transform(fps)としなければいけなかった)。
from rdkit import Chem from rdkit.Chem import AllChem, DataStructs from sklearn.decomposition import PCA from sklearn.cluster import AffinityPropagation from ggplot import * import numpy as np import pandas as pd suppl = Chem.SDMolSupplier('syk.sdf') fps = [] for mol in suppl: fp = AllChem.GetMorganFingerprintAsBitVect(mol, 2) arr = np.zeros((1,)) DataStructs.ConvertToNumpyArray(fp, arr) fps.append(arr) print len(fps) pca = PCA(n_components=2) x = pca.fit(fps).transform(fps) af = AffinityPropagation().fit(x) d = pd.DataFrame(x) d.columns = ["PCA1", "PCA2"] d["labels"] = pd.Series(af.labels_) g = ggplot(aes(x="PCA1", y="PCA2", color="labels"), data=d) + geom_point() + xlab("PCA1") + ylab("PCA2") ggsave("ap.png", g)

 角交換四間飛車 徹底ガイド (マイナビ将棋BOOKS)
角交換四間飛車 徹底ガイド (マイナビ将棋BOOKS) ひと目の中飛車
ひと目の中飛車 ごきげん中飛車を指しこなす本 (最強将棋塾)
ごきげん中飛車を指しこなす本 (最強将棋塾) MEW FIN ミューフィン (ホワイト) (MS)
MEW FIN ミューフィン (ホワイト) (MS) Life Cycle
Life Cycle 久保の石田流
久保の石田流 よくわかる石田流
よくわかる石田流 四間飛車を指しこなす本〈1〉 (最強将棋塾)
四間飛車を指しこなす本〈1〉 (最強将棋塾)