よく分かる中飛車という本を読んでいて、後手ゴキゲン中飛車で相手が7八金型の時に石田流に組み替えろという説明(p. 146)があって、数時間以上悩んでしまった。
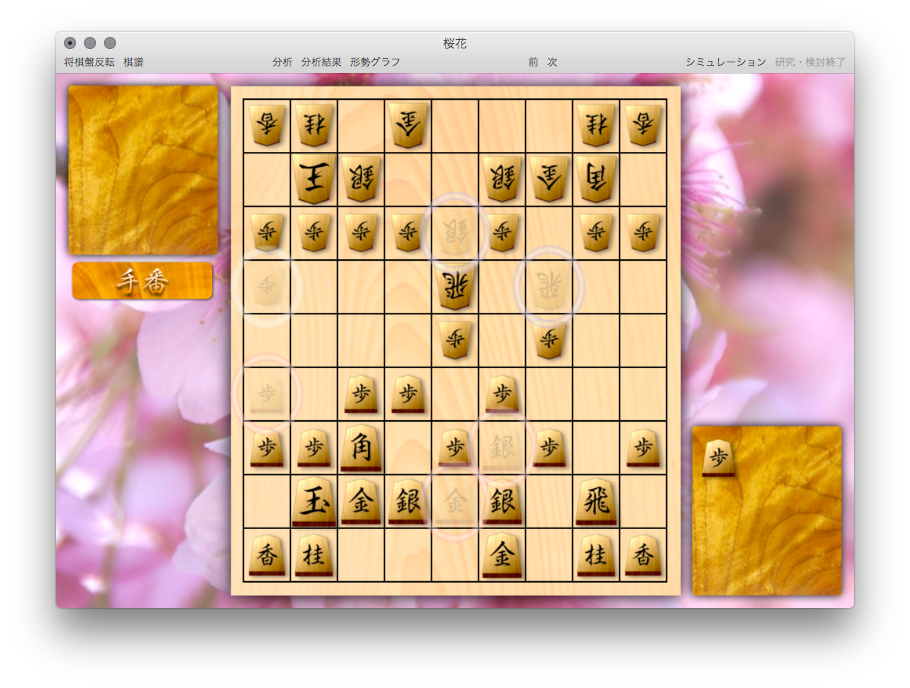
この局面で△2四飛 ▲2五歩 △3四飛と進行するらしいんだが、歩を打ってくれなくて△2四飛 ▲同飛 △同歩と飛車交換された後に▲8二飛と自陣飛車打たれたらどうすんの?
桜花使ってBonanza同士で戦わせてみたんだけど後手負けた…
それにしても桜花便利だ。これ入れてから将棋熱が再燃したからな。あとPDCAが回しやすいので負けた将棋は必ず検討するようになった。
ただ、局面図載せるのに棋譜書き出しさせると
▲7六歩 △3四歩 ▲2六歩 △5四歩 ▲2五歩 △5二飛 ▲7八金 △6二玉
▲6九玉 △7二玉 ▲2四歩 △2四歩 ▲2四飛 △3二金 ▲2八飛 △2三歩
▲4八銀 △5五歩 ▲6八銀 △3五歩 ▲7九玉 △5四飛 ▲7七角 △8二玉
▲8八玉 △7二銀 ▲6六歩 △4二銀 ▲4六歩 △9四歩 ▲4七銀 △9五歩
▲6七銀 △5三銀 ▲5八金 △3四飛 ▲5六歩 △同歩 ▲同銀左 △5四銀
みたいな棋譜が出てくるのでそのままでは局面図ジェネレーターに通すことが出来なくて難儀している。今回スクリーンショット貼り付けたけど、750KByteあるしあまりよろしくないので、コンバーター書くか探すかしないといけないなぁ。
桜花で棋譜並べするのも並べたあとの棋譜を保存できて使いまわせるし、理解が深まるのでいいなと思った。
尚、よくわかる中飛車は(ある程度中飛車わかっているヒトが読むと)よくわかる中飛車という本だなと思って最近まで買って積んであった本ですな。
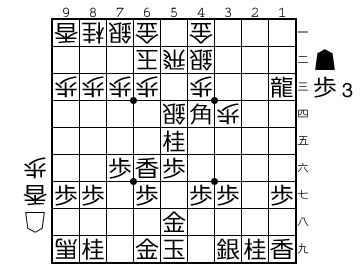

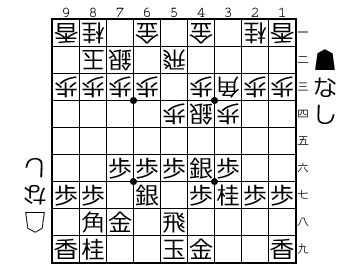

 よくわかる中飛車
よくわかる中飛車






















 ものの歩 1 (ジャンプコミックスDIGITAL)
ものの歩 1 (ジャンプコミックスDIGITAL) 角交換四間飛車 徹底ガイド (マイナビ将棋BOOKS)
角交換四間飛車 徹底ガイド (マイナビ将棋BOOKS) 角交換四間飛車 最新ガイド (マイナビ将棋BOOKS)
角交換四間飛車 最新ガイド (マイナビ将棋BOOKS) マイナビ将棋BOOKS よくわかる石田流
マイナビ将棋BOOKS よくわかる石田流 久保の石田流
久保の石田流





