25102008 chemoinformatics cytoscape
pubchemはクラスタリングの結果をgml形式でダウンロードできるので、それをcytoscapeで読み込んで解析することができる。早速データをとってくる。benzothioleで検索すると大体3000件くらいヒットするのでこれをクラスタリングしたものをgmlでダウンロードして、cytoscapeで読み込んでみた。
適当に描かせて眺めてみる。なんかネットワークっぽくなってますの。
さらに、ズームしてみる。
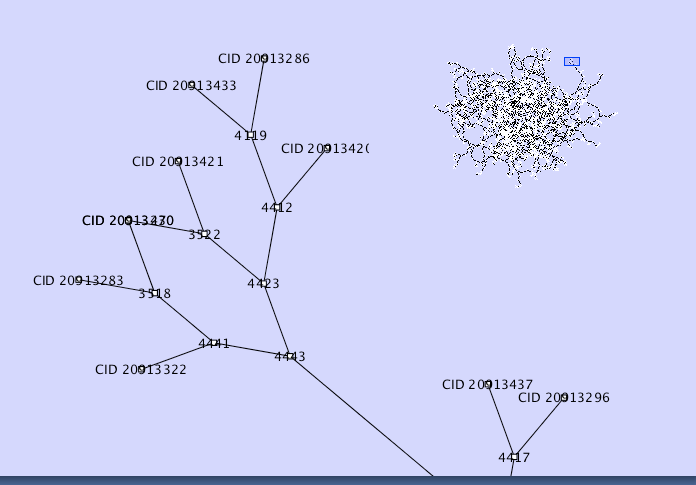
うーん、クラスタリングの結果を見てもちょっと面白くない。むしろ部分構造(MCS:Maximum Common Substructure)から構築したネットワークのほうがわかりやすいだろうな。
あとは、合成の時系列でネットワークをつくればいいと思うが、その場合、元々参考にした化合物の構造への情報をケミストから聞かなきゃいけないのでちょっとめんどいなぁ。
ということはpatentか?あれは先行技術の特許番号ついてたはずだからあれでネットワーク構築すればいいのか。これだったら、描いてみておもしろそう。
TODO: 奇麗な絵が描けるようにcydocなどを参考にちょっと勉強しとく

 サイエンス・ビジネスの挑戦 バイオ産業の失敗の本質を検証する
サイエンス・ビジネスの挑戦 バイオ産業の失敗の本質を検証する グラフィカルモデリング (統計ライブラリー)
グラフィカルモデリング (統計ライブラリー)