コードを記述するコード
evalとか。pythonだとexecか
第二部は「Railsにおけるメタプログラミング」っていう部であまり興味がなかったのでさらっと読み流した。
最後の
メタプログラミングというものなど存在しない。すべてはただのプログラミングじゃ
とこは気に入った。
コードを記述するコード
evalとか。pythonだとexecか
第二部は「Railsにおけるメタプログラミング」っていう部であまり興味がなかったのでさらっと読み流した。
最後の
メタプログラミングというものなど存在しない。すべてはただのプログラミングじゃ
とこは気に入った。
クラス定義
特異クラスとか特異メソッドとか
Rubyの話はなんとなくわかったけど、Pythonで考えた場合にいまいち理解が浅い。
DWの連載あとで読もう
水曜日はブロックの章。
ブロックとは,関数1つを採る高階関数を文法的に特別扱いしただけ,とみなすこともできます。
ほうほう、ということはpythonの場合はラムダ式とればいいのか。
章の終りの方の例がDSLで面白そうなのでやってみる。
def event(name,func):
if func():
print "ALERT: %s" % func.__name__()
event("イベントが発生すること", lambda: True)
event("イベントが発生しないこと", lambda: False)
とやってみたが、ラムダ式は複雑なのが書けないのでコードロック渡したい。
デコレータ使えばいいかということでこんな感じにしてみたが_eventで終わる関数を評価するときに、文字列とって関数適用するところで悩んだ。getattrはまぁわかるとしてimport __main__しないと
Traceback (most recent call last):
File "metr3.py", line 24, in <module>
func = getattr(__main__, func_name)
NameError: name '__main__' is not defined
って出てくる理由がわからん。
def event(eventname):
def decfunc(func):
def func_():
if func():
print "ALERT: %s" % eventname
return func_
return decfunc
@event("イベント発生")
def test1_event(): return True
@event("イベントが発生しないこと")
def test2_event(): return False
if __name__ == '__main__':
import __main__
for func_name in [elem for elem in dir() if elem.endswith("_event")]:
func = getattr(__main__, func_name)
func()
火曜日はメソッドを動的に定義する。Pythonだと__setattr__と__getattribute__を使えばいいが、魔術っぽさを感じる。Rubyは見た目自然じゃないか。
import re
class Ds(object):
def get_cpu_info(self,i):
return 2.16
def get_cpu_price(self,i):
return 150
def get_mouse_info(self,i):
return "dual optical"
def get_mouse_price(self,i):
return 40
# METAPROGRAMMING RUBY
# class computer
# def initialize(computer_id, data_source)
# @id = computer_id
# @data_source = data_source
# data_source.methods.grep(/^get_(.*)_info$/) { Computer.define_compornent $1}
# end
#
# def self.define_component(name)
# define_method(name){
# info = data_source.send "get_#{name}_info", @id
# price = data_source.send "get_#{name}_price", @id
# result = "#{name.capitalize}: #{info} ($#{price})"
# return "* #{result}" if price >= 100
# result
# }
# end
# end
class Computer(object):
def __init__(self,computer_id, data_source):
self.data_source = data_source
self.id = id
for name in [re.search("^get_(.*)_info$",m).group(1) for m in
dir(data_source) if re.search("^get_(.*)_info$",m)]:
self.define_compornent(name)
def define_compornent(self, name):
def define_method():
info = self.data_source.__getattribute__("get_%s_info"%name)(self.id)
price = self.data_source.__getattribute__("get_%s_price"%name)(self.id)
result = "%s: %s ($%s)" % (name.capitalize(), info, price)
if price >= 100:
return "* %s" % result
self.__setattr__(name,define_method)
if __name__ == '__main__':
ds = Ds()
com = Computer(12,ds)
print com.mouse()
print com.cpu()
後半はmethod_missingを使う例だった。perlだとAUTOLOADかとおもってググッてみたら。一覧になってた。
ところで、__getattribute__と__getattr__の違いがわからんという新たな発見をした。
メタプログラミングRubyを読み始めた。第一部は月曜から金曜にわかれていて、、、、、 まぁ毎日読めってことですな。
というわけで、機能はRubyのオブジェクトモデルに関して読んでいた。
rubyにはancestorsっていうメソッドがあって継承関係を調べることができる。
pythonだとこんな感じか?
def ancestors(ins): return [c.__name__ for c in ins.__class__.mro()]
クラスに関してはPython Types and Objectsが分かりやすかった。
あと、pythonでのメタプログラミングはエキスパートPythonで少し触れられている。
rst2pdf で reStructuredText から PDF を生成するで日本語pdfが生成できるようになってもSphinxでは
[ERROR] pdfbuilder.py:120 BuildEnvironment instance has no attribute 'modules'
....
if self.config.pdf_use_modindex and self.env.modules:
AttributeError: BuildEnvironment instance has no attribute 'modules'
FAILED
とかいうエラーが出る(Sphinx1.02,1.03 + rst2pdf-0.15)
この場合にはrst2pdfの新しい版をsvnでインストール(0.16.dev-r2311をいれた)
あとはSphinxで日本語PDFを生成するの通りにやればうまくいく
中身は全然進んでないのに、出力だけは色々できるようになった ;-)
24082010 Python processing
18082010 chemoinformatics Python network igraph
MCSを求めるには比較したい二つの化合物のModular Productを求めて、それの最大クリーク探索をすればいい。
Modular Productとは
にエッジを張ったグラフ。
これを作ってあとはigraphの最大クリーク探索メソッドで
import openbabel as ob
from igraph import *
sm1 = 'OCC(N)=O'
sm2 = 'O=CC(N)=O'
obc = ob.OBConversion()
obc.SetInAndOutFormats('smi','smi')
source = ob.OBMol()
obc.ReadString(source,sm1)
source_atoms = [a for a in ob.OBMolAtomIter(source)]
target = ob.OBMol()
obc.ReadString(target,sm2)
target_atoms = [a for a in ob.OBMolAtomIter(target)]
pairs = []
for i in range(len(source_atoms)):
for j in range(len(target_atoms)):
if source_atoms[i].GetAtomicNum() == target_atoms[j].GetAtomicNum():
pairs.append((source_atoms[i].GetIdx(),target_atoms[j].GetIdx()))
p = []
for x in range(len(pairs)-1):
for y in range(x+1,len(pairs)):
u = pairs[x]
v = pairs[y]
if u[0] != v[0] and u[1] != v[1]:
sb = source.GetBond(u[0],v[0])
tb = target.GetBond(u[1],v[1])
if sb != None: sbo = sb.GetBondOrder()
if tb != None: tbo = tb.GetBondOrder()
if (sb == None and tb == None) or (sb != None and tb != None and sbo == tbo):
p.append((pairs.index(u),pairs.index(v)))
g = Graph(p)
mc = g.largest_cliques()
for c in mc:
print [pairs[i] for i in c]
実際にはline graph(エッジとノードを入れ替えたグラフ)のModular Productをもとめてクリーク探索したほうが計算量がかなり減るらしい。
が、今回はやっていない。
 Handbook of Chemoinformatics Algorithms (Chapman & Hall/Crc Mathematical and Computational Biology)
Handbook of Chemoinformatics Algorithms (Chapman & Hall/Crc Mathematical and Computational Biology) Chemoinformatics and Computational Chemical Biology (Methods in Molecular Biology)
Chemoinformatics and Computational Chemical Biology (Methods in Molecular Biology)16082010 chemoinformatics Python cytoscape
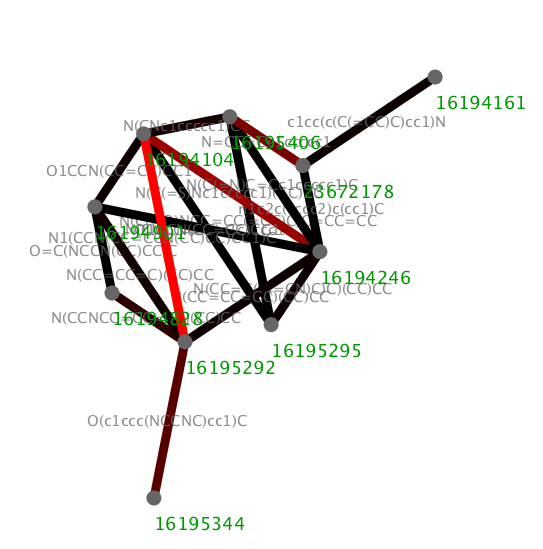
MCSSTanimotoで組んだネットワークのメリットはなんといってもエッジの属性にMCSの情報(smilesとかInChi)をのっけられることであろう。
import sys
from Gaston import MCSSTanimoto,Gaston
import openbabel as ob
obc = ob.OBConversion()
obc.SetInAndOutFormats("sdf", "smi")
input = "pc_sample.sdf"
mol = ob.OBMol()
next = obc.ReadFile(mol,input)
mols = [mol]
while next:
mol.StripSalts(14)
mols.append(mol)
mol = ob.OBMol()
next = obc.Read(mol)
mols = mols[1:11]
for i in range(len(mols)-1):
for j in range(i+1,len(mols)):
sys.stdout.flush()
mcs = MCSSTanimoto(mols[i],mols[j])
if mcs.score() > 0.2:
print "%s\t%s\t%s\t%s\t%2.3f" % (mols[i].GetTitle(), "mcs", \
mols[j].GetTitle(), obc.WriteString(mcs.mcs).split()[0], mcs.score())
でも、実際に組んでみたら、解釈が意外に難しそうなことに気がついた。MCSSTanimotoのアルゴリズム自体が問題なのかもしれない。
MCSよりはmolblaster的に細切れにしてみて最頻出のフラグメントからネットワークを組んでいくか、ありがち置換基の定義済みテーブルを使って類似性の高い置換基動どうしをつなぐという経験ベースのアプローチのほうが直感的かもしれないなぁ。
14082010 Python
SWIGでgastonのPythonバインディング作るのがいいのかもしれないけど、SWIG力が足りないので、外部コマンドから使っている。
os.environ['PATH']で探してみたけど、普通はどうやるんだろうか
if 'gaston' not in reduce(lambda a,b: a+b,[os.listdir(d) for d in \
os.environ['PATH'].split(':') if os.path.isdir(d)]):
print "gaston: command not found"
exit()
os.system("gaston %d %s %s > /dev/null 2>&1" % (freq,gasfile,output))