ココのエントリでの構造描画は流れ的にChemrubyで描くべきなんだけど。
そもそも画像描画のステップには
- 2次元座標を保持したオブジェクトの生成
- 画像描画
が必要で、smilesのような座標情報のない形での構造情報は、何かで構造立ち上げしないといけない。chemrubyは立ち上げ処理できないらしいので、この作業は他の何かに任せるわけだ。 ちなみに、perlmolは芳香環が含まれない場合には、座標のついた形でmol形式に保存してくれるんだが、座標があったりなかったりとかはかなり使いにくいし、普通芳香環のない構造扱うことってあんまないでしょ?
な感じなので、CDKを使うのがデフォルト。
で、CDKで"OC=C-C=C"をmol形式にするとこうなる
CDK
5 4 0 0 0 0 0 0 0 0999 V2000
0.0000 0.0000 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2990 -0.7500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5981 -0.0000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8971 -0.7500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1962 -0.0000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2 1 1 0 0 0 0
3 2 2 0 0 0 0
4 3 1 0 0 0 0
5 4 2 0 0 0 0
M END
うまく座標が割り当てられてますな。これをchemrubyを使ってpngに変換するとこうなる
irb(main):001:0> require 'RMagick'
=> true
irb(main):002:0> require 'chem'
=> true
irb(main):003:0> mol = Chem.open_mol("diene.mol")
irb(main):004:0> mol.save("rbtest.png")
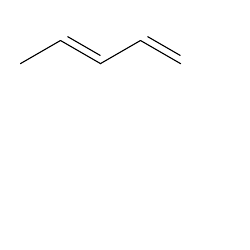
水酸基の情報が落ちてしまったヨ。
