07 08 2010 chemoinformatics R Python ggplot2 Tweet
昨日の化合物の類似性ネットワークのやつは何がメリットなのかなぁと考えてみたけど、一つにはedgeの数を自由に決められるあたりなんじゃないかと。あんまり低い閾値だとなんでもかんでもつながって何見てるかわからんし、逆に厳しい閾値にしちゃうとシングルトンばかりになっちゃうし。
というわけで、プロット欠かせてそれ見て閾値決める必要はあるんだろうなと。最終的にはeddgesの数とか、ノードあたりに生えているedgeの数の平均とかを統計的に処理して自動で閾値を決めるのが楽なんだろうけど、、、(一般的な方法あるのかな?)。X-meansに倣って、BICとかAIC使ってみようかなぁ。
で、プロット描いた。最初に書いたコードがナイーブすぎて遅くていらっときたので書き直した。まだかなり遅いけど、耐えられるのでこれでよしとした。
import pybel
mols = list(pybel.readfile("sdf", "pc_sample.sdf"))
fps = [x.calcfp() for x in mols]
similarity_matrix = []
for i in range(len(fps)):
for j in range(i+1,len(fps)):
similarity = fps[i] | fps[j]
similarity_matrix.append(similarity)
threshold = 0.0
print "threshold\tnodes"
while threshold < 100:
t = threshold / 100.0
new_matrix = [e for e in similarity_matrix if e > t]
nodes = len(new_matrix)
print "%2.1f\t%d" % (threshold, nodes)
similarity_matrix = new_matrix
threshold += 0.1
あとはRで
library(ggplot2)
nodes <- read.table("xxxx",header=T)
qplot(threshold,edgess, data=nodes, log="y")
dev.copy(device=png,filename="xxxxxx")
dev.off()
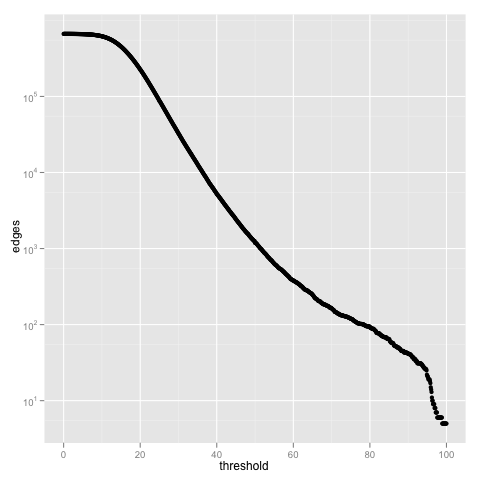
node数が1158なのでedgeの数が10000から100あたりの間でちょうどいい閾値を探したいんだけど、これだけじゃわからん。やっぱシングルトンの数とかノードあたりに生えているエッジ数の平均とか数えないとだめかな。
