Caco-2細胞のモデリング
設問1
Vmax は結合サイト数、ATPターンオーバー、細胞総数からもとまる
> 100000*1e+6*25/6.02e+23*60
[1] 2.491694e-10
つまり2.491694(nmol/min)
library(odesolve)
params = c(Vmax=2.491694*10^-3,S=4.71,Pa=5e-6,Pb=3e-6,Km=0.3,Va=1.5,Vc=0.025,Vb=3.0)
times=c(0,(1:60))
dydt <- function(t,y,p){
Vmax <- p['Vmax']
S <- p['S']
Pa <- p['Pa']
Pb <- p['Pb']
Km <- p['Km']
Va <- p['Va']
Vc <- p['Vc']
Vb <- p['Vb']
Ca <- (S*Pa*(y[2]-y[1]) + Vmax*y[2]/(Km+y[2]))/Va
Cc <- (S*(Pa*(y[1]-y[2]) + Pb*(y[3]-y[2])) - Vmax*y[2]/(Km+y[2]))/Vc
Cb <- (S*Pb*(y[2]-y[3]))/Vb
return(list(c(Ca,Cc,Cb)))
}
濃度リストをあたえるとPeffのリストを返す関数を書いておく。
calc <- function(dose_list){
l <- c()
for (dose in dose_list){
y <- lsoda(c(Ca=dose,Cc=0.0,Cb=0.0),times,dydt,params)
Y <- data.frame(y)
res <- lm(Cb ~ time,Y[c("time","Cb")][seq(41,61),])
Peff <- res$coefficients[2]*3/(4.71*dose)
l <- append(l,Peff)
}
return(l)
}
d <- c(0.1,0.3,0.6,1,3,6,10,30,60,100,300,600,900,1000)
peffs <- calc(d)
plot(d,peffs)
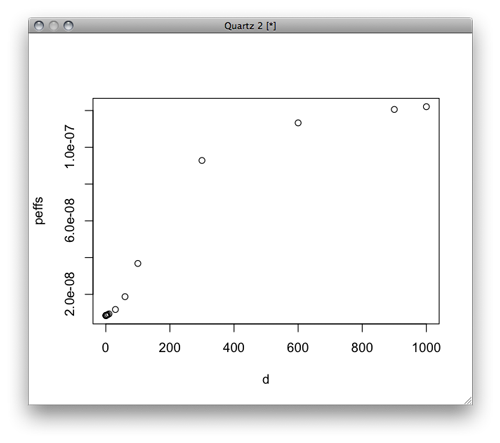
Pgpが飽和した後(高濃度側)ではシグモイド。
設問2
省略

 ファーマコキネティクス―演習による理解
ファーマコキネティクス―演習による理解