Ruby on Railsで、遺伝子発現データベースを!というエントリがすんげー面白かったので、ここはひとつperlなcatalystでやってみたヨ。
ichan::Weblog - Ruby on Rails Gruffを使って、11分で作る遺伝子発現データベース
ごく簡単な遺伝子発現データベースをRuby on Rails で作ってみました。時系列の遺伝子発現データを取りこんで、Gruffで折れ線グラフで表示します。bioinformaticsやマイクロアレイ解析と関係がないかたもGruffの使用例として参考になると思います。
一応、10分でできるRoRなやつをトレースしたことがある。なんで、今回ムービーも見ながら、できるだけ同じような感じで構築してみた。ちょっとCatalystとRoRの対比をしておきますと、
RoRはレールにのっかってスイスイッと。特にDBまわりとの連携がすごく楽。対してCatalystはページングの処理とかプラグイン使わないといけないし、フォームの用意も自前でやらないとあかん。 画像吐き出しはRoRはコントローラで処理しているが、CatalystはViewに押し込んでいる。GD使うつもりだったけどTTでGD呼ぶのは抵抗があったので、Catalystのプラグイン探したけどなかった。そんなわけで結局、画像処理はTTのGD::Graphプラグインで行なってます。
個人的な見解だけど、RoRは定型的な処理は楽ちんっぽいです。CatalystだとページングにClass::DBI::Sweet使うかClass::DBI::Pager使うか悩んだり、Class::DBI::Sweetのヘルパーないかと探し回ったりと、コードの長さの割りに時間がかかってしまった。でも、viewに画像処理押し込むとコントローラーいじらなくてすむのでそれはそれでいいのかな?
さて手動かし履歴(かなり端折ってますが)
まず、発現データの処理とDB化は元ネタサイトどおりにbioconductorで。スクリプト動かしたら、パッケージ足りなかったので
source("http://www.bioconductor.org/biocLite.R") biocLite(c("mgu74av2cdf"))
を。あと、Rでapllyを使うと早いのね、勉強になった。今度他のデータで試してみよう。
$ catalyst.pl Expview $ cd Expview $ ./script/expview_create.pl view TT TT $ script/expview_create.pl model CDBI CDBI dbi:SQLite:myexp
これだと、ページングの処理が出来ないので、lib/Expview/Model/CDBI.pmに
use Class::DBI::Pager;
を追加。
そしてコントローラーまわりをいじる。lib/Expview/Cotroller/Root.pmを編集
sub list : Global { my ( $self, $c ) = @_; my $page = $c->request->param('page') || 1; my $pager = Expview::Model::CDBI::Expressions->pager(10,$page); $c->stash->{items} = $pager->retrieve_all; $c->stash->{prev_page} = $page-1; $c->stash->{next_page} = $page+1; $c->stash->{template} = "templates/view.tt"; $c->forward('Expview::View::TT'); } sub graph : Global { my ( $self, $c ) = @_; my $id = $c->request->param('id'); my $exp = Expview::Model::CDBI::Expressions->retrieve($id); my @expression = ($exp->d0,$exp->d2,$exp->d4,$exp->d10); $c->stash->{exp} = \@expression; $c->stash->{affyid} = $exp->affyid; $c->stash->{template} = "templates/graph.tt"; $c->forward('Expview::View::TT'); }
こんな感じで、リスト用のルーチンと画像表示用のルーチンを記述。TT用のテンプレートもroot/templatesに用意します。
これでOK
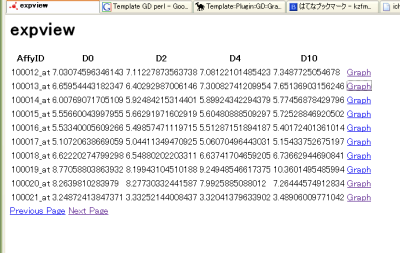
リストはこんな感じで表示されますが、Catalystは自前でテンプレート書かないといけないのに対して、RoRはそこも面倒見てくれている。この部分のコストはお気軽さに結構きいてくるかも。
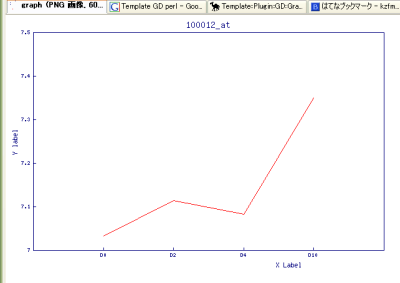
画像もGD::Graphで。ちょっと見栄えがよくないが、でもテンプレートいじれば綺麗になるヨ。
今回作った、expview.tar.gzをダウンロードできるようにしておきました。
$ tar xvfz expview.tar.gz $ cd Expview $ ./script/expview_server.pl
でport3000番にアクセスすればいいはず。
5/15追記
Catalyst::EnzymeというCatalyst用CRUDフレームワークを使えば、テンプレートとかCRUDまわりをまとめてやってくれるので、10分くらいでできそうな感じデス。
