CatalystとGD::Graph::linesで作った遺伝子発現データベースでCRUDメンドイとかいたたがCatalyst::EnzymeというCatalyst用CRUDフレームワークがあったので使ってみた。
CatalystにEnzymeなんて、バイオインフォ+ケモインフォな僕にはソソル名前のフレームワークだヨ。
$ catalyst.pl Eview
$ cd Eview/
$ ./script/eview_create.pl view TT Enzyme::TT
$ ./script/eview_create.pl model MyExp Enzyme::CDBI dbi:SQLite:dbname=db/myexp.db
$ ./script/eview_create.pl controller Expressions Enzyme::CRUD MyExp::Expressions
とかやって、テストサーバー立ち上げるとこんな感じで。
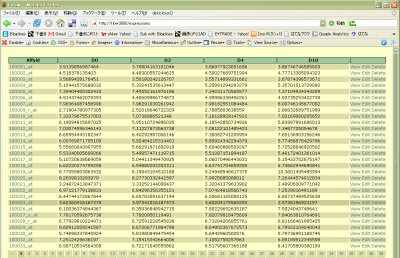
かなり、楽チンだ。
Catalyst::Enzyme - Catalyst用CRUDフレームワーク
前提条件アプリケーションがClass::DBIベースのモデル・クラスを使っていること。
アプリケーションの一部として(たぶん内部用の管理インタフェースとして)、あるいは単に何かのとっかかりとしてCRUD機能を使いたいと思っていること。
(コードにしろテンプレートにしろ)修正するためにソースを読んで理解しようとする気構えがあること。Enzymeを使えばいままでより速く、また少ないコード量で始めることはできますが、その後、自分自身の用途にあわせたフレームワークへと作り込んでいく必要はあるのです。自分の必要に応じてどんどん変更していってください。
特に最後に書いてあるように、ソース読めないと作りこみではまるかもしれん。
