chemoinformatics的には最もlateなフェーズである、QSPkRとかモデル動物からヒト外挿あたりにブームがやってきたので、色々と調べたりいじったりする日々を。
どっちかっていうとPK/PD,PBPKのようなモデルに興味があるのでそういったパッケージがないかと、CRANを漁った。
CRAN Task View: Analysis of Pharmacokinetic DataによるとPK/PD modelはnlmeODEパッケージを使えばいいらしい。
PBPKに関してはR-ForgeにRDynamicってのを見つけた。
ちょっとnlmeODEのサンプルを
data(Theoph)
TheophODE <- Theoph
TheophODE$Dose[TheophODE$Time!=0] <- 0
TheophODE$Cmt <- rep(1,dim(TheophODE)[1])
OneComp <- list(DiffEq=list(
dy1dt = ~ -ka*y1 ,dy2dt = ~ ka*y1-ke*y2),
ObsEq=list(
c1 = ~ 0,
c2 = ~ y2/CL*ke),
Parms=c("ka","ke","CL"),
States=c("y1","y2"),
Init=list(0,0))
TheophModel <- nlmeODE(OneComp,TheophODE)
Theoph.nlme <- nlme(conc ~ TheophModel(ka,ke,CL,Time,Subject),
data = TheophODE, fixed=ka+ke+CL~1, random = pdDiag(ka+CL~1),
start=c(ka=0.5,ke=-2.5,CL=-3.2),
control=list(returnObject=TRUE,msVerbose=TRUE),
verbose=TRUE)
plot(augPred(Theoph.nlme,level=0:1))
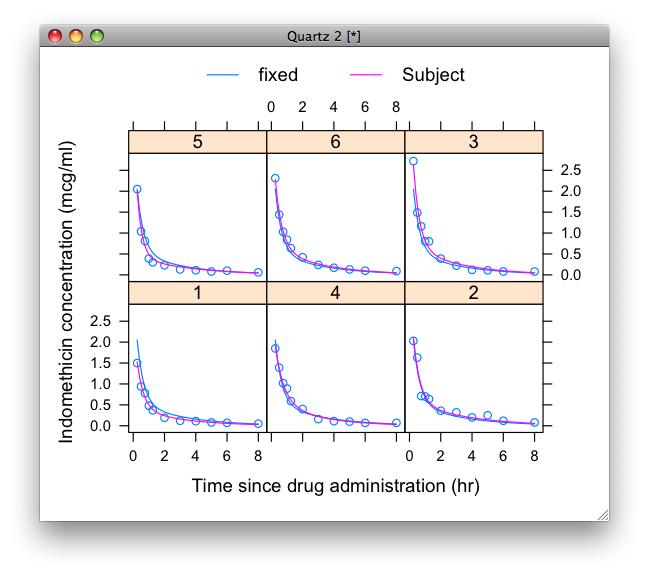
よくよく考えてみるに、既に3年くらい前にはこういうことに必要性を認識していたのだけどなぁ。

 薬物代謝学―医療薬学・毒性学の基礎として
薬物代謝学―医療薬学・毒性学の基礎として
 Computer Applications in Pharmaceutical Research and Development (Wiley Series in Drug Discovery and Development)
Computer Applications in Pharmaceutical Research and Development (Wiley Series in Drug Discovery and Development)