08 07 2014 chemoinformatics Python Tweet
K-meansのように予めクラスタ数を指定すると、「そのクラスタ数は正しいの?」っていう疑問が浮かぶと思う。
「なんらかの統計値に基づいて適切なクラスタに分割して欲しい」そんな願いを叶えるのがAffinity Propagationというクラスタリングアルゴリズムである
exemplara(セントロイドとかクラスタ中心)になるべきパラメータ(responsibility)とクラスタメンバに属しやすさ(availability)を交互に更新していって収束させる手法なので、K-meansのような初期値依存性がないらしい。
クラスタ数は類似度行列の対角要素(自分との類似度)に依存する(デフォルトはmedian)のでここを変更するとクラスタ数も変わるんだけどね。
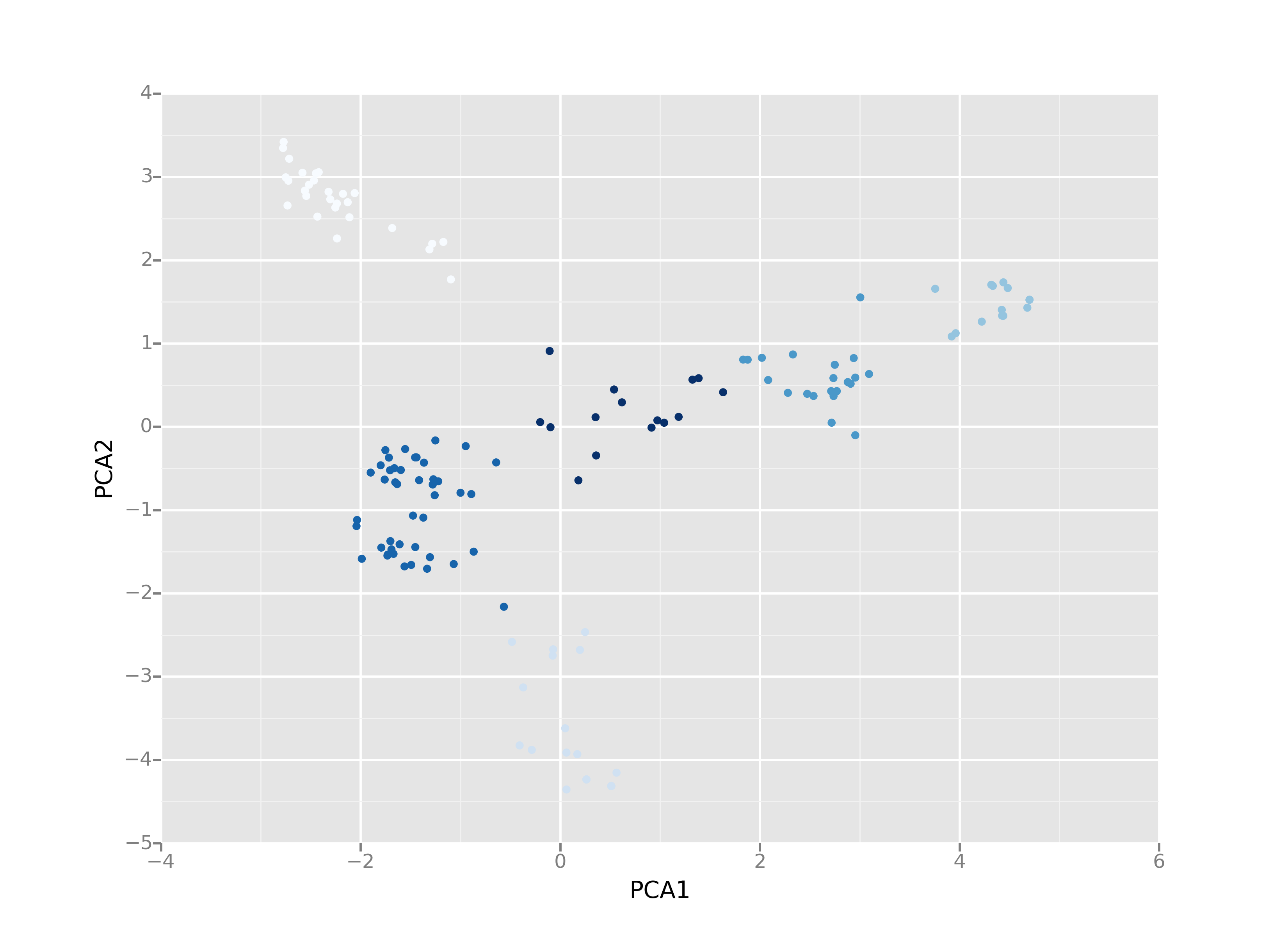
Scikit-learnではAffinity Propagationが実装されているのでsykのケミストリースペースを作ってクラスタリングしてみた。ちなみにスライドのPCAの説明は間違っていた(pca.fit(fps).transform(fps)としなければいけなかった)。
from rdkit import Chem from rdkit.Chem import AllChem, DataStructs from sklearn.decomposition import PCA from sklearn.cluster import AffinityPropagation from ggplot import * import numpy as np import pandas as pd suppl = Chem.SDMolSupplier('syk.sdf') fps = [] for mol in suppl: fp = AllChem.GetMorganFingerprintAsBitVect(mol, 2) arr = np.zeros((1,)) DataStructs.ConvertToNumpyArray(fp, arr) fps.append(arr) print len(fps) pca = PCA(n_components=2) x = pca.fit(fps).transform(fps) af = AffinityPropagation().fit(x) d = pd.DataFrame(x) d.columns = ["PCA1", "PCA2"] d["labels"] = pd.Series(af.labels_) g = ggplot(aes(x="PCA1", y="PCA2", color="labels"), data=d) + geom_point() + xlab("PCA1") + ylab("PCA2") ggsave("ap.png", g)